Gene
KWMTBOMO16837
Annotation
PREDICTED:_dentin_sialophosphoprotein-like_[Biomphalaria_glabrata]
Location in the cell
Nuclear Reliability : 2.198
Sequence
CDS
ATGTTGACAATTACCGTCTATGTAATAATACAGTGTAATGTAATCGTTTCTTTTGTTACAGCATCTGCCGCATCCACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAATCCTACTTCAGATCCAAGTGCGTCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAATGCATCTGCCGCATCCACGACCGATACATCTGTAACGGATAAACCCACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGGTACATCTGTAACGGATAAACCTACTTCAGATCAAAGTGCATCTGCCCGCATCGACGACGATACATCTGTAACGGATAATCCTACTTCAGATCCAAGTGCATCTGCCGCGTCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCGTCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAATCCTACTTCAGATCCAAGTGCGTCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCAAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCGTCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCGTCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAATCCTACTTCAGATCCAAGTGCGTCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACCCATACATCTGTAACGGATAAACCTACTTCAGATCAAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCGTCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATATATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCGTCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCACATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATATATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCGTCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATATATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCGTCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCGTCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCGACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCCAAGTGTAAGTTTCCATAAAAAGGTTGACAATTACCGTCTATCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACCGATACATCTGTAACGGATAAACCTACTTCAGATCAAAGTGCATCTACCGCATCGACGACCGATACATCTGTAACGGACAAACCTACTTCAGATCCAAGTGCATCTGCCGCATCCACGACAGACAAATCTGTAACGGATAAACCTACTTCAGATCCAAGTGGTAAGTTTCCATAA
Protein
MLTITVYVIIQCNVIVSFVTASAASTTDTSVTDKPTSDPSASAASTTDTSVTDKPTSDPSASAASTTDTSVTDNPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPNASAASTTDTSVTDKPTSDPSASAASTTGTSVTDKPTSDQSASARIDDDTSVTDNPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDNPTSDPSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTDTSVTDKPTSDQSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDNPTSDPSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTHTSVTDKPTSDQSASTASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDISVTDKPTSDPSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASATSTTDKSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDISVTDKPTSDPSASAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDISVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSASAASTTDTSVTDKPTSDPSVSFHKKVDNYRLSSAASTTDKSVTDKPTSDPSASTASTTDTSVTDKPTSDPSASAASTTDTSVTDKPTSDQSASTASTTDTSVTDKPTSDPSASAASTTDKSVTDKPTSDPSGKFP
Summary
Uniprot
ProteinModelPortal
Ontologies
GO
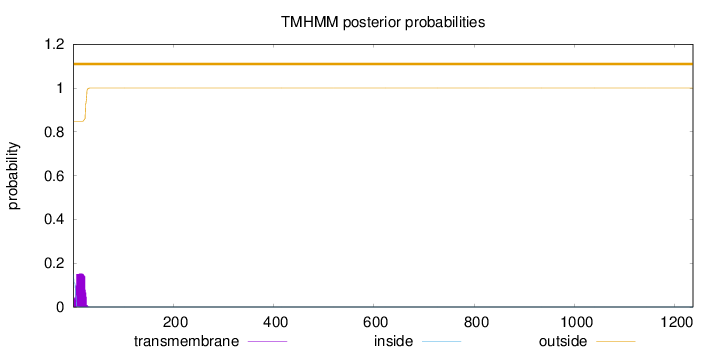
Topology
SignalP
Position: 1 - 23,
Likelihood: 0.516210
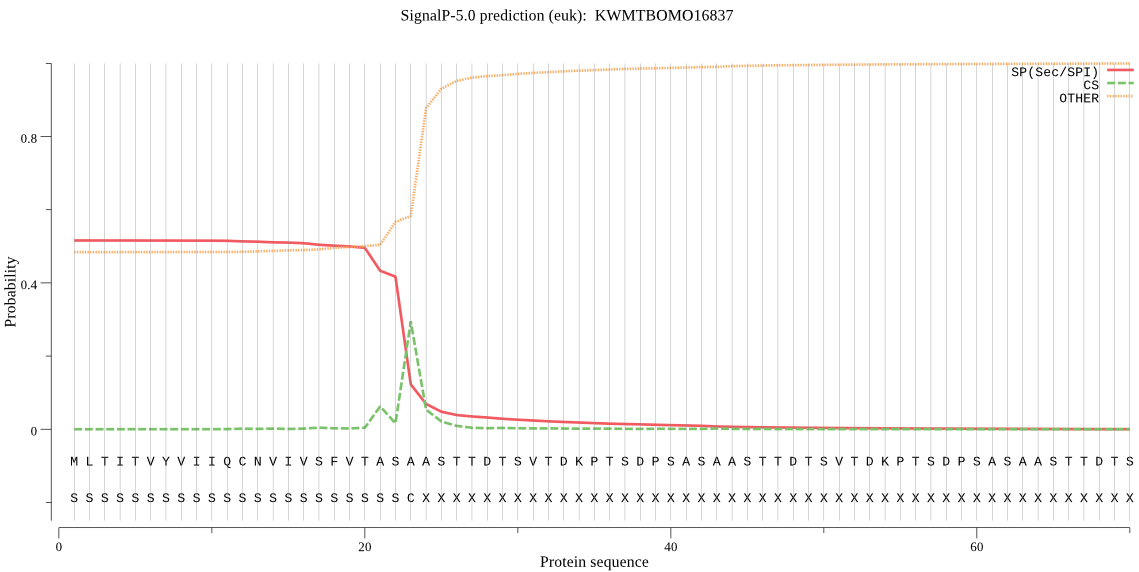
Length:
1236
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
3.22767
Exp number, first 60 AAs:
3.22767
Total prob of N-in:
0.15199
outside
1 - 1236
Population Genetic Test Statistics
Pi
0
Theta
0
Tajima's D
0
CLR
0
CSRT
0
Interpretation
Uncertain
