Gene
KWMTBOMO14053
Pre Gene Modal
BGIBMGA011233
Annotation
PREDICTED:_zinc_finger_protein_845-like_[Bombyx_mori]
Location in the cell
Nuclear Reliability : 4.545
Sequence
CDS
ATGATAGTCGAGCATAAATGTACGGAGACTGGAGAGAAAATAATTACCCCCCTCAAATGTGCTTTGTGCGATTACACAACACTTTTCAAGGACAATCTGAACGCTCACAAGCGAAAACACAAGTCCGAAAAGCGTTACCAATGTGAGAATTGTACATATTCAACTTCGTACATCAACAATTATAAGAAACACAAAAAGAAGCATTCGAAAGAAATTGTCGAATTTAAATGTGACAAATGTTCGTTTGTAACAAAACATTCAGGTCACATACACAGGCATCTGTCTCAGATACATAACGAGATAACGGAGAAAGCAAAGAAATGTGAATTTTGTGATTTTACAACAATAGTTGGATGGAGATTGAACATACACAAGCAAAGGAGTCGCCAGAATGAAGTCATAAAGTGCGTTTACTGTGAATACCAAACTTTTTATAAATGCGAAAGCAAGAAACACAGTAATACACATTTCAAGGAAATCTACACGAAAAACTCACATAGAAATAAACATAATAATATTCAAGAACAAAATGTAGACGAAACTTTGAGTTTGAATCTCGGTGAGCAAAACCTTCCTTCAATCACGAATCAAACGAACCACAGCTCATACAATATCAACTCCTATACGAATACCAGTAATTACAGTCAAAACTATTCGAACGCTGAAGATTTTTTACCGAAAGAACTGTTAAATTACACGGAGATATCAAATGAATTACCGTTGACTTACAGCACCCCATATTCAGATAAAATCGACAAGAAAACCATTCAAGTATTGGACTCTAATGACAGAGAAAGACCATTTGGTTGTTCTCATTGCAATTACATATCGAAATTCAAGGCAACGGTGCAGAGGCACTATCAAAGACACCACACTGACGACAAGCGCCCGTACCACTGCTCCAACTGCGACTTCAAAACGAAGACCAAAGATCAGATAGCGTTGCACAACAAACGCAGCGCCTCATCCCTAGAAATGAGATGTCAGAATTGTGATTTCACAACGAATTTCAAATGCCAATTCGTTATGCATCAGCGCTCCCACTACACGTTCAAATGTGAACTGTGTAATTACACGTGTAAGCATAAGCATGAGATCGAGAAGCACGTGAGGACGATTCACATGGGCACAAAGCACCTGTGCAAGTATTGCGACTTCAAAACTCCTAAATTGGAAGCGCTGCTGTGTCACGAAGCATTGCACACCGGCAACAAACCGTTTAAATGCAAGTTGTGTGACTACCAGACTGTGAGGCTCTCCCTTTTGGACATACACGTCAGGAGGTACCACAGTGATTTGAAGGTAGTCAAACTTATTAGCGAGAGTAAAGTGCAATCCCTAAAGGTATCCGTTCCACCTGATGAATGTTCGTGA
Protein
MIVEHKCTETGEKIITPLKCALCDYTTLFKDNLNAHKRKHKSEKRYQCENCTYSTSYINNYKKHKKKHSKEIVEFKCDKCSFVTKHSGHIHRHLSQIHNEITEKAKKCEFCDFTTIVGWRLNIHKQRSRQNEVIKCVYCEYQTFYKCESKKHSNTHFKEIYTKNSHRNKHNNIQEQNVDETLSLNLGEQNLPSITNQTNHSSYNINSYTNTSNYSQNYSNAEDFLPKELLNYTEISNELPLTYSTPYSDKIDKKTIQVLDSNDRERPFGCSHCNYISKFKATVQRHYQRHHTDDKRPYHCSNCDFKTKTKDQIALHNKRSASSLEMRCQNCDFTTNFKCQFVMHQRSHYTFKCELCNYTCKHKHEIEKHVRTIHMGTKHLCKYCDFKTPKLEALLCHEALHTGNKPFKCKLCDYQTVRLSLLDIHVRRYHSDLKVVKLISESKVQSLKVSVPPDECS
Summary
Uniprot
H9JNX9
A0A212FGI1
A0A2A4IYE9
A0A2H1VP67
A0A3S2NXL0
A0A0L7KM65
+ More
A0A1E1WNY7 C4A0A2 C3YHM6 C3YHQ7 C3ZRC8 C3ZSM4 C3Z3I8 C4A0A1 C3YMF4 C3YZZ2 C3ZXA9 C3XX14 C3Y7F9 C3ZY73 C3Z011 C3YU42 C3YXW0 A0A2R7X7B9 C3YWR8 C3ZYL3 C3YHJ7 A0A0V0G4M8 C3ZRC6 C3XX02 C3YNB1 C3YHK1 C3YWR0 C3XXB0 C3Z309 C3ZXA8 C3YHM7 C3XSV2 A0A023F6Z3 C3YXV5 C3YXW3 C3ZR95 A0A1B6DQK6 C3YP61 C3XWZ0 A0A1B6DVS6 C3YHR4 C3Z6C9 C3YHY0 C3YKM0 C3Z007 C3YBB2 C3YKT3 C3ZRA1 C3Z658 C3Z3H3 C3YQG4 C3YU40 C3YGC5 C3YHN3 A0A1B6CRV2 C4A0Z6 C3XSP6 C3ZR99 C3ZZW5 C3YU97 C3Z009 C3YQQ3 C3YPH0 C3XWY0 C3XQN9 A0A069DVZ9 C3ZWS5 C4A0F8 C3YMG4 C3YCB1 A0A0P4VSQ8 A0A023F0S8 C3XX19 C3ZRB5 C3ZE44 C4A0A4 C3ZGY2 A0A224XS64 A0A224XNK1 C3XXA8
A0A1E1WNY7 C4A0A2 C3YHM6 C3YHQ7 C3ZRC8 C3ZSM4 C3Z3I8 C4A0A1 C3YMF4 C3YZZ2 C3ZXA9 C3XX14 C3Y7F9 C3ZY73 C3Z011 C3YU42 C3YXW0 A0A2R7X7B9 C3YWR8 C3ZYL3 C3YHJ7 A0A0V0G4M8 C3ZRC6 C3XX02 C3YNB1 C3YHK1 C3YWR0 C3XXB0 C3Z309 C3ZXA8 C3YHM7 C3XSV2 A0A023F6Z3 C3YXV5 C3YXW3 C3ZR95 A0A1B6DQK6 C3YP61 C3XWZ0 A0A1B6DVS6 C3YHR4 C3Z6C9 C3YHY0 C3YKM0 C3Z007 C3YBB2 C3YKT3 C3ZRA1 C3Z658 C3Z3H3 C3YQG4 C3YU40 C3YGC5 C3YHN3 A0A1B6CRV2 C4A0Z6 C3XSP6 C3ZR99 C3ZZW5 C3YU97 C3Z009 C3YQQ3 C3YPH0 C3XWY0 C3XQN9 A0A069DVZ9 C3ZWS5 C4A0F8 C3YMG4 C3YCB1 A0A0P4VSQ8 A0A023F0S8 C3XX19 C3ZRB5 C3ZE44 C4A0A4 C3ZGY2 A0A224XS64 A0A224XNK1 C3XXA8
EMBL
BABH01009777
BABH01009778
AGBW02008661
OWR52844.1
NWSH01005461
PCG64200.1
+ More
ODYU01003613 SOQ42596.1 RSAL01000031 RVE51577.1 JTDY01008776 KOB64407.1 GDQN01002334 JAT88720.1 GG666795 EEN41783.1 GG666514 EEN60013.1 EEN60044.1 GG666664 EEN45001.1 GG666674 EEN44426.1 GG666576 EEN53032.1 EEN41782.1 GG666529 EEN58748.1 GG666567 EEN54051.1 GG666710 EEN42789.1 GG666471 EEN67274.1 GG666489 EEN63787.1 GG666722 EEN42504.1 EEN54070.1 GG666552 EEN56319.1 GG666563 EEN54919.1 KK858243 PTY27688.1 GG666561 EEN55312.1 GG666732 EEN42360.1 EEN59984.1 GECL01003123 JAP03001.1 EEN44999.1 EEN67262.1 GG666533 EEN58217.1 EEN59988.1 EEN55304.1 EEN67370.1 GG666575 EEN53048.1 EEN42788.1 EEN60014.1 GG666459 EEN69023.1 GBBI01001743 JAC16969.1 EEN54914.1 EEN54922.1 EEN44968.1 GEDC01009332 JAS27966.1 GG666537 EEN57952.1 EEN67250.1 GEDC01007526 JAS29772.1 EEN60051.1 GG666586 EEN51904.1 EEN60117.1 GG666523 EEN59088.1 EEN54066.1 GG666497 EEN62269.1 EEN59151.1 EEN44974.1 GG666584 EEN51984.1 EEN53017.1 GG666542 EEN57494.1 EEN56317.1 GG666511 EEN60646.1 EEN60020.1 GEDC01021201 JAS16097.1 GG666840 EEN41537.1 EEN68967.1 EEN44972.1 GG666770 EEN41918.1 EEN56374.1 EEN54068.1 GG666543 EEN57409.1 GG666538 EEN57754.1 EEN67240.1 GG666455 EEN69615.1 GBGD01000878 JAC88011.1 GG666703 EEN42994.1 GG666803 EEN41719.1 EEN58758.1 GG666500 EEN62143.1 GDKW01003599 JAI52996.1 GBBI01003632 JAC15080.1 EEN67279.1 EEN44988.1 GG666612 EEN49000.1 EEN41785.1 GG666621 EEN48129.1 GFTR01005585 JAW10841.1 GFTR01006264 JAW10162.1 EEN67368.1
ODYU01003613 SOQ42596.1 RSAL01000031 RVE51577.1 JTDY01008776 KOB64407.1 GDQN01002334 JAT88720.1 GG666795 EEN41783.1 GG666514 EEN60013.1 EEN60044.1 GG666664 EEN45001.1 GG666674 EEN44426.1 GG666576 EEN53032.1 EEN41782.1 GG666529 EEN58748.1 GG666567 EEN54051.1 GG666710 EEN42789.1 GG666471 EEN67274.1 GG666489 EEN63787.1 GG666722 EEN42504.1 EEN54070.1 GG666552 EEN56319.1 GG666563 EEN54919.1 KK858243 PTY27688.1 GG666561 EEN55312.1 GG666732 EEN42360.1 EEN59984.1 GECL01003123 JAP03001.1 EEN44999.1 EEN67262.1 GG666533 EEN58217.1 EEN59988.1 EEN55304.1 EEN67370.1 GG666575 EEN53048.1 EEN42788.1 EEN60014.1 GG666459 EEN69023.1 GBBI01001743 JAC16969.1 EEN54914.1 EEN54922.1 EEN44968.1 GEDC01009332 JAS27966.1 GG666537 EEN57952.1 EEN67250.1 GEDC01007526 JAS29772.1 EEN60051.1 GG666586 EEN51904.1 EEN60117.1 GG666523 EEN59088.1 EEN54066.1 GG666497 EEN62269.1 EEN59151.1 EEN44974.1 GG666584 EEN51984.1 EEN53017.1 GG666542 EEN57494.1 EEN56317.1 GG666511 EEN60646.1 EEN60020.1 GEDC01021201 JAS16097.1 GG666840 EEN41537.1 EEN68967.1 EEN44972.1 GG666770 EEN41918.1 EEN56374.1 EEN54068.1 GG666543 EEN57409.1 GG666538 EEN57754.1 EEN67240.1 GG666455 EEN69615.1 GBGD01000878 JAC88011.1 GG666703 EEN42994.1 GG666803 EEN41719.1 EEN58758.1 GG666500 EEN62143.1 GDKW01003599 JAI52996.1 GBBI01003632 JAC15080.1 EEN67279.1 EEN44988.1 GG666612 EEN49000.1 EEN41785.1 GG666621 EEN48129.1 GFTR01005585 JAW10841.1 GFTR01006264 JAW10162.1 EEN67368.1
Proteomes
PRIDE
Pfam
PF00096 zf-C2H2
Interpro
SUPFAM
SSF57667
SSF57667
ProteinModelPortal
H9JNX9
A0A212FGI1
A0A2A4IYE9
A0A2H1VP67
A0A3S2NXL0
A0A0L7KM65
+ More
A0A1E1WNY7 C4A0A2 C3YHM6 C3YHQ7 C3ZRC8 C3ZSM4 C3Z3I8 C4A0A1 C3YMF4 C3YZZ2 C3ZXA9 C3XX14 C3Y7F9 C3ZY73 C3Z011 C3YU42 C3YXW0 A0A2R7X7B9 C3YWR8 C3ZYL3 C3YHJ7 A0A0V0G4M8 C3ZRC6 C3XX02 C3YNB1 C3YHK1 C3YWR0 C3XXB0 C3Z309 C3ZXA8 C3YHM7 C3XSV2 A0A023F6Z3 C3YXV5 C3YXW3 C3ZR95 A0A1B6DQK6 C3YP61 C3XWZ0 A0A1B6DVS6 C3YHR4 C3Z6C9 C3YHY0 C3YKM0 C3Z007 C3YBB2 C3YKT3 C3ZRA1 C3Z658 C3Z3H3 C3YQG4 C3YU40 C3YGC5 C3YHN3 A0A1B6CRV2 C4A0Z6 C3XSP6 C3ZR99 C3ZZW5 C3YU97 C3Z009 C3YQQ3 C3YPH0 C3XWY0 C3XQN9 A0A069DVZ9 C3ZWS5 C4A0F8 C3YMG4 C3YCB1 A0A0P4VSQ8 A0A023F0S8 C3XX19 C3ZRB5 C3ZE44 C4A0A4 C3ZGY2 A0A224XS64 A0A224XNK1 C3XXA8
A0A1E1WNY7 C4A0A2 C3YHM6 C3YHQ7 C3ZRC8 C3ZSM4 C3Z3I8 C4A0A1 C3YMF4 C3YZZ2 C3ZXA9 C3XX14 C3Y7F9 C3ZY73 C3Z011 C3YU42 C3YXW0 A0A2R7X7B9 C3YWR8 C3ZYL3 C3YHJ7 A0A0V0G4M8 C3ZRC6 C3XX02 C3YNB1 C3YHK1 C3YWR0 C3XXB0 C3Z309 C3ZXA8 C3YHM7 C3XSV2 A0A023F6Z3 C3YXV5 C3YXW3 C3ZR95 A0A1B6DQK6 C3YP61 C3XWZ0 A0A1B6DVS6 C3YHR4 C3Z6C9 C3YHY0 C3YKM0 C3Z007 C3YBB2 C3YKT3 C3ZRA1 C3Z658 C3Z3H3 C3YQG4 C3YU40 C3YGC5 C3YHN3 A0A1B6CRV2 C4A0Z6 C3XSP6 C3ZR99 C3ZZW5 C3YU97 C3Z009 C3YQQ3 C3YPH0 C3XWY0 C3XQN9 A0A069DVZ9 C3ZWS5 C4A0F8 C3YMG4 C3YCB1 A0A0P4VSQ8 A0A023F0S8 C3XX19 C3ZRB5 C3ZE44 C4A0A4 C3ZGY2 A0A224XS64 A0A224XNK1 C3XXA8
PDB
5UND
E-value=1.49238e-12,
Score=177
Ontologies
GO
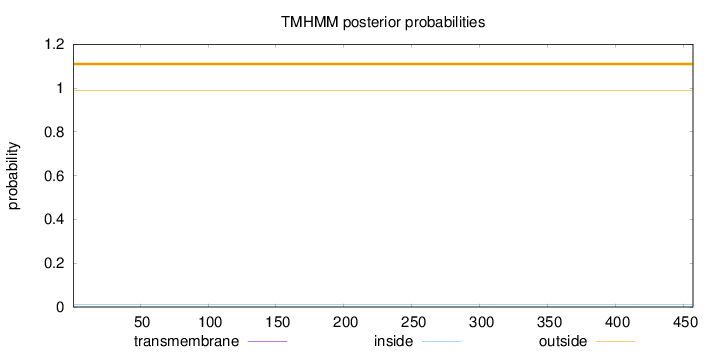
Topology
Length:
457
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
0.00018
Exp number, first 60 AAs:
0.00018
Total prob of N-in:
0.01038
outside
1 - 457
Population Genetic Test Statistics
Pi
170.841425
Theta
194.435952
Tajima's D
-0.446178
CLR
0.941599
CSRT
0.255687215639218
Interpretation
Uncertain
