Gene
KWMTBOMO13182
Pre Gene Modal
BGIBMGA000353
Annotation
beta-1?3-glucan_recognition_protein_4_precursor_[Bombyx_mori]
Location in the cell
Extracellular Reliability : 2.733
Sequence
CDS
ATGTGGCTGTTAACTCTGGGCGTAGTTGCCCTAATATCGGCGAGTAAAGCTTGCACACCCAGCGTAACTACGGTGAGCGGTACTCACGCCCCTGTGACCGTGTGTTCCGGACAACTCATTTTTGCTGATGACTTCGTCGACTTCGACCTTGAGAAATGGCAGCACGAAAACACCTTAGCCGGAGGTGGTAACTGGGAGTTCCAGTACTACAACAACAATAGAACTAACTCATTCACGAACAACGGTCTACTGTACATTCGGCCGTCCCTTACGTCCGACCAGTTCGGATCCGCATTTTTACATAGCGGTCGCTTGAACATTGAAGGAGGCGCACCCGCCGACAGATGCACTAACCCTCAATGGTACGGTTGCGAGCGTGTTGGTACCCCCACTAATATTTTGAACCCCATTAAAAGTGCACGTATTCGCACCGTTCATTCCTTCAGCTTCCAGTATGGTAAAGTAGAAGTTAGAGCGAAAATGCCTTCCGGAGATTGGCTCTGGCCCGCAATTTGGTTGATGCCTGCATACAACAAATACGGCACCTGGCCAGCTTCTGGGGAAATCGACCTTGTCGAATCCAGAGGCAACAAAAACATGTTCCTCAACGGTCTACACATTGGTACACAGGAAGCTGGTTCGACACTGCACTACGGACCTTTCCCCGGACTAAGCGGATGGGAAAAAGCGCACTGGGTCAGAAGGAACAGCGCCGGATACGACACTAATTTCCATCGTTACCAACTGGAATGGACACCAGATTTCATAAGCTTTAGAATTGACGACTCTGAGATCGGTCGTGTCACTCCTGGAAATGGTGGCTTTTGGGAATACGGCGGCTTCAACAATAGACCCGGTATTCATAACCCGTGGAGATATGGAAGTAAGATGGCGCCATTCGACCAAAAATTCTATCTCATAATCAACTTAGCTGTCGGAGGCACCAATGGTTTCTTCCCTGATGGTGTCAAGAACCCTATTCCTAAGCCTTGGTGGAACAATTCCCCAATGGCAGCCACAGATTTCTGGAATGGCCAAGGCGGTTGGCTTCCGACGTGGAATTTGAACGTTAACGATGGTCAAGACGCTTCCCTCCAAGTAGACTACGTTCGAGTTTGGGCGTTATAA
Protein
MWLLTLGVVALISASKACTPSVTTVSGTHAPVTVCSGQLIFADDFVDFDLEKWQHENTLAGGGNWEFQYYNNNRTNSFTNNGLLYIRPSLTSDQFGSAFLHSGRLNIEGGAPADRCTNPQWYGCERVGTPTNILNPIKSARIRTVHSFSFQYGKVEVRAKMPSGDWLWPAIWLMPAYNKYGTWPASGEIDLVESRGNKNMFLNGLHIGTQEAGSTLHYGPFPGLSGWEKAHWVRRNSAGYDTNFHRYQLEWTPDFISFRIDDSEIGRVTPGNGGFWEYGGFNNRPGIHNPWRYGSKMAPFDQKFYLIINLAVGGTNGFFPDGVKNPIPKPWWNNSPMAATDFWNGQGGWLPTWNLNVNDGQDASLQVDYVRVWAL
Summary
Uniprot
H9ISX5
C8C9T4
G9G7R0
G9G7R2
A0A194R0W3
A0A194PY35
+ More
G9G7R1 B6CMG2 B1NLE1 B7SVK5 A0A2H1W0K3 B7SVM3 B7SVL8 A6YQV6 A0A3S2NK35 A6YQV7 B7SVK7 B7SVL9 A0A212EV85 B7SVL1 G1BJ40 Q2N3Z7 Q2N3X7 Q2N3Y1 Q2N3Y4 J9RVF6 Q2N3Y3 Q2N3Z3 Q2N3Z4 Q2N3Y8 Q2N3Z5 Q2N3Y7 Q2N3X6 Q2N3Y9 H8YU82 Q2N3Z1 J9RDQ2 Q2N3Y0 Q2N3Z0 Q2N3Z6 Q2N3X8 Q2N3X4 Q2N3Z2 A0A067RDX6 A0A2J7QLK6 Q2N3X5 G1BJ39 A0A1V1G500 Q2N3X9 Q2N3Y2 J9S505 G1BJ37 D9D797 L0CMF6 A0A067RBH9 A0A336MV15 A0A1B0D0K8 Q2N3X2 Q2N3X3 A0A2J7QLL7 D9D798 D9D7A2 D9D7C3 A0A3G4ZJN2 D9D7A7 D9D7A0 D9D7B4 D9D7A6 D9D799 D9D7B7 J9S7T1 D9D7D0 D9D7B8 D9D7B2 D9D7A8 G1BJ35 D9D7D1 D9D7C0 D9D7D2 D9D7B6 Q2N3Y5 A0A182N794 A0A084VE22 A0A182HFF2 Q2N3Y6 A0A182G107 A0A1D8MC22 Q7QCT6 A0A1D8MC28 A0A1D8MC25 A0A1D8MC33 A0A1D8MC41 A0A1D8MC54 A0A1D8MC67 A0A1D8MC31 A0A1D8MC42 A0A1D8MC30 A0A1D8MBZ7 A0A1D8MC44
G9G7R1 B6CMG2 B1NLE1 B7SVK5 A0A2H1W0K3 B7SVM3 B7SVL8 A6YQV6 A0A3S2NK35 A6YQV7 B7SVK7 B7SVL9 A0A212EV85 B7SVL1 G1BJ40 Q2N3Z7 Q2N3X7 Q2N3Y1 Q2N3Y4 J9RVF6 Q2N3Y3 Q2N3Z3 Q2N3Z4 Q2N3Y8 Q2N3Z5 Q2N3Y7 Q2N3X6 Q2N3Y9 H8YU82 Q2N3Z1 J9RDQ2 Q2N3Y0 Q2N3Z0 Q2N3Z6 Q2N3X8 Q2N3X4 Q2N3Z2 A0A067RDX6 A0A2J7QLK6 Q2N3X5 G1BJ39 A0A1V1G500 Q2N3X9 Q2N3Y2 J9S505 G1BJ37 D9D797 L0CMF6 A0A067RBH9 A0A336MV15 A0A1B0D0K8 Q2N3X2 Q2N3X3 A0A2J7QLL7 D9D798 D9D7A2 D9D7C3 A0A3G4ZJN2 D9D7A7 D9D7A0 D9D7B4 D9D7A6 D9D799 D9D7B7 J9S7T1 D9D7D0 D9D7B8 D9D7B2 D9D7A8 G1BJ35 D9D7D1 D9D7C0 D9D7D2 D9D7B6 Q2N3Y5 A0A182N794 A0A084VE22 A0A182HFF2 Q2N3Y6 A0A182G107 A0A1D8MC22 Q7QCT6 A0A1D8MC28 A0A1D8MC25 A0A1D8MC33 A0A1D8MC41 A0A1D8MC54 A0A1D8MC67 A0A1D8MC31 A0A1D8MC42 A0A1D8MC30 A0A1D8MBZ7 A0A1D8MC44
Pubmed
EMBL
BABH01013028
GQ372969
ACU57045.1
JN248786
AEV66276.1
JN248788
+ More
AEV66278.1 KQ460883 KPJ11342.1 KQ459586 KPI97938.1 JN248787 AEV66277.1 EU325562 ACB54952.1 EF600056 ABU98621.1 EU770374 ACI32818.1 ODYU01005529 SOQ46466.1 EU770392 ACI32836.1 EU770387 ACI32831.1 EF641300 ABR28478.2 RSAL01000387 RVE42024.1 EF641301 ABR28479.1 EU770376 ACI32820.1 EU770388 ACI32832.1 AGBW02012225 OWR45410.1 EU770380 ACI32824.1 JF683378 AEK64801.1 DQ058910 AAZ08480.1 DQ058930 AAZ08500.1 DQ058926 AAZ08496.1 DQ058923 AAZ08493.1 JQ435783 AFR46661.1 DQ058924 AAZ08494.1 DQ058914 AAZ08484.1 DQ058913 AAZ08483.1 DQ058919 AAZ08489.1 DQ058912 AAZ08482.1 DQ058920 AAZ08490.1 DQ058931 AAZ08501.1 DQ058918 AAZ08488.1 JF915524 AFD54026.1 DQ058916 AAZ08486.1 JQ435784 AFR46662.1 DQ058927 AAZ08497.1 DQ058917 AAZ08487.1 DQ058911 AAZ08481.1 DQ058929 AAZ08499.1 DQ058933 AAZ08503.1 DQ058915 AAZ08485.1 KK852567 KDR21213.1 NEVH01013243 PNF29475.1 DQ058932 AAZ08502.1 JF683377 AEK64800.1 FX985263 BAX07276.1 DQ058928 AAZ08498.1 DQ058925 AAZ08495.1 JQ435786 AFR46664.1 JF683375 AEK64798.1 GU906821 ADJ19004.1 JX876645 AGA16572.1 KDR21211.1 UFQT01002820 SSX34106.1 AJVK01021487 AJVK01021488 AJVK01021489 AJVK01021490 AJVK01021491 DQ058935 AAZ08505.1 DQ058934 AAZ08504.1 PNF29473.1 GU906822 GU906833 GU906834 GU906837 ADJ19005.1 ADJ19016.1 ADJ19017.1 ADJ19020.1 GU906826 GU906835 GU906839 ADJ19009.1 ADJ19018.1 ADJ19022.1 GU906847 GU906848 GU906851 GU906852 GU906853 ADJ19030.1 ADJ19031.1 ADJ19034.1 ADJ19035.1 ADJ19036.1 MH669347 AYV75052.1 GU906831 ADJ19014.1 GU906824 GU906825 GU906827 GU906828 GU906829 ADJ19007.1 ADJ19008.1 ADJ19010.1 ADJ19011.1 ADJ19012.1 GU906838 ADJ19021.1 GU906830 ADJ19013.1 GU906823 ADJ19006.1 GU906841 ADJ19024.1 JQ435785 AFR46663.1 GU906854 ADJ19037.1 GU906842 GU906843 ADJ19025.1 ADJ19026.1 GU906836 ADJ19019.1 GU906832 ADJ19015.1 JF683373 AEK64796.1 GU906855 ADJ19038.1 GU906844 GU906845 GU906846 GU906849 GU906850 ADJ19027.1 ADJ19028.1 ADJ19029.1 ADJ19032.1 ADJ19033.1 GU906856 JF683376 ADJ19039.1 AEK64799.1 GU906840 ADJ19023.1 DQ058922 AAZ08492.1 ATLV01012167 KE524768 KFB36216.1 JXUM01037026 KQ561114 KXJ79663.1 DQ058921 AAZ08491.1 JXUM01037825 KQ561143 KXJ79574.1 KX620813 AOV81819.1 AAAB01008859 EAA07705.4 KX620810 AOV81816.1 KX620814 AOV81820.1 KX620825 AOV81831.1 KX620808 AOV81814.1 KX620827 AOV81833.1 KX620836 AOV81842.1 KX620822 AOV81828.1 KX620828 KX620830 KX620833 AOV81834.1 AOV81836.1 AOV81839.1 KX620798 KX620807 KX620819 KX620820 KX620829 KX620835 KX620838 KX620856 AOV81804.1 AOV81813.1 AOV81825.1 AOV81826.1 AOV81835.1 AOV81841.1 AOV81844.1 AOV81862.1 KX620787 KX620790 KX620792 KX620811 KX620834 AOV81793.1 AOV81796.1 AOV81798.1 AOV81817.1 AOV81840.1 KX620806 KX620826 KX620839 AOV81812.1 AOV81832.1 AOV81845.1
AEV66278.1 KQ460883 KPJ11342.1 KQ459586 KPI97938.1 JN248787 AEV66277.1 EU325562 ACB54952.1 EF600056 ABU98621.1 EU770374 ACI32818.1 ODYU01005529 SOQ46466.1 EU770392 ACI32836.1 EU770387 ACI32831.1 EF641300 ABR28478.2 RSAL01000387 RVE42024.1 EF641301 ABR28479.1 EU770376 ACI32820.1 EU770388 ACI32832.1 AGBW02012225 OWR45410.1 EU770380 ACI32824.1 JF683378 AEK64801.1 DQ058910 AAZ08480.1 DQ058930 AAZ08500.1 DQ058926 AAZ08496.1 DQ058923 AAZ08493.1 JQ435783 AFR46661.1 DQ058924 AAZ08494.1 DQ058914 AAZ08484.1 DQ058913 AAZ08483.1 DQ058919 AAZ08489.1 DQ058912 AAZ08482.1 DQ058920 AAZ08490.1 DQ058931 AAZ08501.1 DQ058918 AAZ08488.1 JF915524 AFD54026.1 DQ058916 AAZ08486.1 JQ435784 AFR46662.1 DQ058927 AAZ08497.1 DQ058917 AAZ08487.1 DQ058911 AAZ08481.1 DQ058929 AAZ08499.1 DQ058933 AAZ08503.1 DQ058915 AAZ08485.1 KK852567 KDR21213.1 NEVH01013243 PNF29475.1 DQ058932 AAZ08502.1 JF683377 AEK64800.1 FX985263 BAX07276.1 DQ058928 AAZ08498.1 DQ058925 AAZ08495.1 JQ435786 AFR46664.1 JF683375 AEK64798.1 GU906821 ADJ19004.1 JX876645 AGA16572.1 KDR21211.1 UFQT01002820 SSX34106.1 AJVK01021487 AJVK01021488 AJVK01021489 AJVK01021490 AJVK01021491 DQ058935 AAZ08505.1 DQ058934 AAZ08504.1 PNF29473.1 GU906822 GU906833 GU906834 GU906837 ADJ19005.1 ADJ19016.1 ADJ19017.1 ADJ19020.1 GU906826 GU906835 GU906839 ADJ19009.1 ADJ19018.1 ADJ19022.1 GU906847 GU906848 GU906851 GU906852 GU906853 ADJ19030.1 ADJ19031.1 ADJ19034.1 ADJ19035.1 ADJ19036.1 MH669347 AYV75052.1 GU906831 ADJ19014.1 GU906824 GU906825 GU906827 GU906828 GU906829 ADJ19007.1 ADJ19008.1 ADJ19010.1 ADJ19011.1 ADJ19012.1 GU906838 ADJ19021.1 GU906830 ADJ19013.1 GU906823 ADJ19006.1 GU906841 ADJ19024.1 JQ435785 AFR46663.1 GU906854 ADJ19037.1 GU906842 GU906843 ADJ19025.1 ADJ19026.1 GU906836 ADJ19019.1 GU906832 ADJ19015.1 JF683373 AEK64796.1 GU906855 ADJ19038.1 GU906844 GU906845 GU906846 GU906849 GU906850 ADJ19027.1 ADJ19028.1 ADJ19029.1 ADJ19032.1 ADJ19033.1 GU906856 JF683376 ADJ19039.1 AEK64799.1 GU906840 ADJ19023.1 DQ058922 AAZ08492.1 ATLV01012167 KE524768 KFB36216.1 JXUM01037026 KQ561114 KXJ79663.1 DQ058921 AAZ08491.1 JXUM01037825 KQ561143 KXJ79574.1 KX620813 AOV81819.1 AAAB01008859 EAA07705.4 KX620810 AOV81816.1 KX620814 AOV81820.1 KX620825 AOV81831.1 KX620808 AOV81814.1 KX620827 AOV81833.1 KX620836 AOV81842.1 KX620822 AOV81828.1 KX620828 KX620830 KX620833 AOV81834.1 AOV81836.1 AOV81839.1 KX620798 KX620807 KX620819 KX620820 KX620829 KX620835 KX620838 KX620856 AOV81804.1 AOV81813.1 AOV81825.1 AOV81826.1 AOV81835.1 AOV81841.1 AOV81844.1 AOV81862.1 KX620787 KX620790 KX620792 KX620811 KX620834 AOV81793.1 AOV81796.1 AOV81798.1 AOV81817.1 AOV81840.1 KX620806 KX620826 KX620839 AOV81812.1 AOV81832.1 AOV81845.1
Proteomes
Pfam
PF00722 Glyco_hydro_16
SUPFAM
SSF49899
SSF49899
ProteinModelPortal
H9ISX5
C8C9T4
G9G7R0
G9G7R2
A0A194R0W3
A0A194PY35
+ More
G9G7R1 B6CMG2 B1NLE1 B7SVK5 A0A2H1W0K3 B7SVM3 B7SVL8 A6YQV6 A0A3S2NK35 A6YQV7 B7SVK7 B7SVL9 A0A212EV85 B7SVL1 G1BJ40 Q2N3Z7 Q2N3X7 Q2N3Y1 Q2N3Y4 J9RVF6 Q2N3Y3 Q2N3Z3 Q2N3Z4 Q2N3Y8 Q2N3Z5 Q2N3Y7 Q2N3X6 Q2N3Y9 H8YU82 Q2N3Z1 J9RDQ2 Q2N3Y0 Q2N3Z0 Q2N3Z6 Q2N3X8 Q2N3X4 Q2N3Z2 A0A067RDX6 A0A2J7QLK6 Q2N3X5 G1BJ39 A0A1V1G500 Q2N3X9 Q2N3Y2 J9S505 G1BJ37 D9D797 L0CMF6 A0A067RBH9 A0A336MV15 A0A1B0D0K8 Q2N3X2 Q2N3X3 A0A2J7QLL7 D9D798 D9D7A2 D9D7C3 A0A3G4ZJN2 D9D7A7 D9D7A0 D9D7B4 D9D7A6 D9D799 D9D7B7 J9S7T1 D9D7D0 D9D7B8 D9D7B2 D9D7A8 G1BJ35 D9D7D1 D9D7C0 D9D7D2 D9D7B6 Q2N3Y5 A0A182N794 A0A084VE22 A0A182HFF2 Q2N3Y6 A0A182G107 A0A1D8MC22 Q7QCT6 A0A1D8MC28 A0A1D8MC25 A0A1D8MC33 A0A1D8MC41 A0A1D8MC54 A0A1D8MC67 A0A1D8MC31 A0A1D8MC42 A0A1D8MC30 A0A1D8MBZ7 A0A1D8MC44
G9G7R1 B6CMG2 B1NLE1 B7SVK5 A0A2H1W0K3 B7SVM3 B7SVL8 A6YQV6 A0A3S2NK35 A6YQV7 B7SVK7 B7SVL9 A0A212EV85 B7SVL1 G1BJ40 Q2N3Z7 Q2N3X7 Q2N3Y1 Q2N3Y4 J9RVF6 Q2N3Y3 Q2N3Z3 Q2N3Z4 Q2N3Y8 Q2N3Z5 Q2N3Y7 Q2N3X6 Q2N3Y9 H8YU82 Q2N3Z1 J9RDQ2 Q2N3Y0 Q2N3Z0 Q2N3Z6 Q2N3X8 Q2N3X4 Q2N3Z2 A0A067RDX6 A0A2J7QLK6 Q2N3X5 G1BJ39 A0A1V1G500 Q2N3X9 Q2N3Y2 J9S505 G1BJ37 D9D797 L0CMF6 A0A067RBH9 A0A336MV15 A0A1B0D0K8 Q2N3X2 Q2N3X3 A0A2J7QLL7 D9D798 D9D7A2 D9D7C3 A0A3G4ZJN2 D9D7A7 D9D7A0 D9D7B4 D9D7A6 D9D799 D9D7B7 J9S7T1 D9D7D0 D9D7B8 D9D7B2 D9D7A8 G1BJ35 D9D7D1 D9D7C0 D9D7D2 D9D7B6 Q2N3Y5 A0A182N794 A0A084VE22 A0A182HFF2 Q2N3Y6 A0A182G107 A0A1D8MC22 Q7QCT6 A0A1D8MC28 A0A1D8MC25 A0A1D8MC33 A0A1D8MC41 A0A1D8MC54 A0A1D8MC67 A0A1D8MC31 A0A1D8MC42 A0A1D8MC30 A0A1D8MBZ7 A0A1D8MC44
PDB
3ILN
E-value=1.8833e-18,
Score=227
Ontologies
GO
Topology
SignalP
Position: 1 - 17,
Likelihood: 0.996315
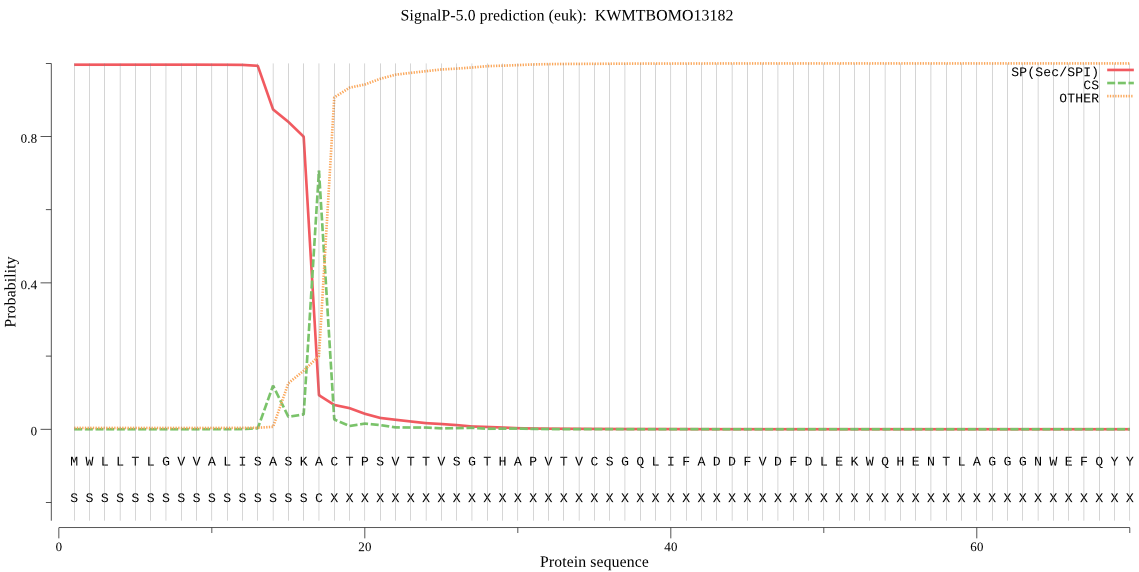
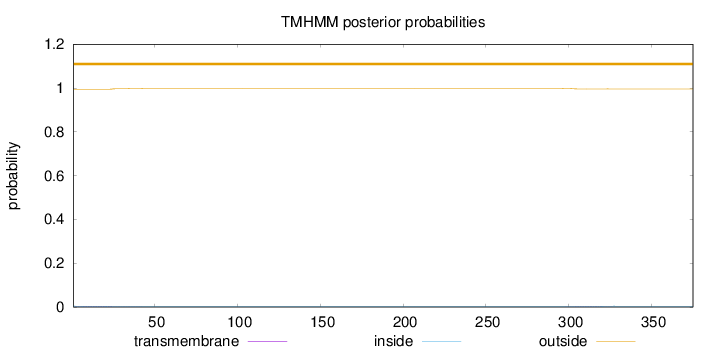
Length:
375
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
0.10613
Exp number, first 60 AAs:
0.0649
Total prob of N-in:
0.00529
outside
1 - 375
Population Genetic Test Statistics
Pi
200.615551
Theta
188.788345
Tajima's D
0.196418
CLR
0.09103
CSRT
0.426778661066947
Interpretation
Uncertain
