Pre Gene Modal
BGIBMGA000231
Annotation
vacuolar_ATP_synthase_21_kDa_proteolipid_subunit_[Bombyx_mori]
Location in the cell
PlasmaMembrane Reliability : 4.237
Sequence
CDS
ATGAGATACTTCTTAAGTTACTTATTCGTGCTCCTGGTGGGACTTGCAATCCCGATATTTTCGCTGTACTACGTCCTCAATGGAAAGGGTGAGCAGATAAGTTTGGGATGGTTCTTGGAGAACACTTCCCCGTATATGTGGGGTACCCTCGGAATCGCCTTTTCCGTTGCCCTGTCCGTTGTCGGAGCAGCCATGGGCATCCACACTACTGGTGTCAGCATAGTGGGAGGAGGTGTCAAAGCACCTAGAATCAAGACTAAGAATTTGATCTCCGTCATCTTCTGCGAGGCCGTCGCCATTTACGGTTTGATCACTGCTATCGTGCTCTCTGGTATGCTGGAGAAATATTCTGAACCATTTACTAGTGTGTCCGTCAAGCAGCAGAACTGGATGGCGGGATACGTGATGTTCGGCGCTGGACTCGCTGTTGGCTTGGTGAATCTATTCTGTGGAATTGCTGTTGGTATCGTGGGCTCTGGTGCTGCTCTAGCGGATGCTGCCAACGCTGCCTTGTTCGTCAAGATCCTCATCGTTGAGATTTTTGGATCTGCCATTGGTCTTTTTGGCCTTATTGTTGGCATTTATATGACATCAAAAGTAAAAATGGGCAACCAGTAA
Protein
MRYFLSYLFVLLVGLAIPIFSLYYVLNGKGEQISLGWFLENTSPYMWGTLGIAFSVALSVVGAAMGIHTTGVSIVGGGVKAPRIKTKNLISVIFCEAVAIYGLITAIVLSGMLEKYSEPFTSVSVKQQNWMAGYVMFGAGLAVGLVNLFCGIAVGIVGSGAALADAANAALFVKILIVEIFGSAIGLFGLIVGIYMTSKVKMGNQ
Summary
Similarity
Belongs to the V-ATPase proteolipid subunit family.
Uniprot
Q2F630
A0A0N0PBL6
A0A194Q852
A0A0C5DCC5
A0A0C5DGH6
A0A0C5DBS7
+ More
A0A0C5DCF6 A0A0C5DU88 A0A0C5DBW0 A0A0C5D7T1 A0A0C5DUC0 A0A0C5DBU1 A0A0C5D7R7 A0A0C5D7R1 A0A0C5DBT1 A0A0C5D7S5 A0A0C5DCF3 A0A0C5DU86 A0A0C5DGJ6 A0A0C5DGJ7 A0A0C5DBT3 A0A0C5DU73 A0A0C5DCE0 A0A0C5DGM6 A0A0C5D7W4 A0A0C5DU61 A0A0C5DCD2 A0A0C5D7Q3 A0A0C5DBX3 A0A0C5DBR5 A0A0C5DGH5 A0A0C5D7X7 A0A0C5D7W9 A0A0C5DCD8 A0A0C5D7V7 A0A0C5DBT9 A0A0C5DGI3 A0A0C5DUB3 A0A0C5DU68 A0A0C5D7U6 A0A0C5DGL3 A0A0C5D7V3 A0A0C5DCD4 A0A0C5DBV1 A0A0C5DU94 A0A0C5DCJ3 A0A0C5DUA2 A0A0C5DBU8 A0A0C5DCI0 A0A0C5D7U8 A0A0C5DUD0 A0A0C5DCJ7 A0A0C5DCG5 A0A0C5DGL5 A0A0C5DGI9 A0A0C5D7T6 A0A0C5DBT6 A0A0C5DUC5 A0A0C5DU76 A0A0C5DUA6 A0A0C5DGM0 A0A0C5DU83 S4PYG2 A0A0L7LH25 A0A0C5DBQ7 A0A0C5DU55 A0A0C5DGN3 A0A0C5DCK2 A0A0C5D7T9 A0A0C5D7S1 A0A0C5D7U2 A0A0C5DCE7 A0A0C5DCF9 A0A0C5DGJ4 A0A0C5DUB7 A0A0C5DGG7 A0A0C5DGL8 A0A2A4J6K8 A0A2H1WIH9 A0A0C5DU51 A0A1E1W7P6 A0A0C5DU79 G3K8G5 G3K8E0 G3K8D7 A0A0C5D7P6 A0A0C5D7S8 G3K8E2 G3K8G8 G3K8E7 G3K8G4 A0A0C5DGH1 A0A3S2NYT8 A0A0C5DBR1 A0A0C5D7R3 A0A0C5DGH9 A0A0C5DCC3 A0A0C5DU59 A0A0C5DCI8 A0A0C5DCI4
A0A0C5DCF6 A0A0C5DU88 A0A0C5DBW0 A0A0C5D7T1 A0A0C5DUC0 A0A0C5DBU1 A0A0C5D7R7 A0A0C5D7R1 A0A0C5DBT1 A0A0C5D7S5 A0A0C5DCF3 A0A0C5DU86 A0A0C5DGJ6 A0A0C5DGJ7 A0A0C5DBT3 A0A0C5DU73 A0A0C5DCE0 A0A0C5DGM6 A0A0C5D7W4 A0A0C5DU61 A0A0C5DCD2 A0A0C5D7Q3 A0A0C5DBX3 A0A0C5DBR5 A0A0C5DGH5 A0A0C5D7X7 A0A0C5D7W9 A0A0C5DCD8 A0A0C5D7V7 A0A0C5DBT9 A0A0C5DGI3 A0A0C5DUB3 A0A0C5DU68 A0A0C5D7U6 A0A0C5DGL3 A0A0C5D7V3 A0A0C5DCD4 A0A0C5DBV1 A0A0C5DU94 A0A0C5DCJ3 A0A0C5DUA2 A0A0C5DBU8 A0A0C5DCI0 A0A0C5D7U8 A0A0C5DUD0 A0A0C5DCJ7 A0A0C5DCG5 A0A0C5DGL5 A0A0C5DGI9 A0A0C5D7T6 A0A0C5DBT6 A0A0C5DUC5 A0A0C5DU76 A0A0C5DUA6 A0A0C5DGM0 A0A0C5DU83 S4PYG2 A0A0L7LH25 A0A0C5DBQ7 A0A0C5DU55 A0A0C5DGN3 A0A0C5DCK2 A0A0C5D7T9 A0A0C5D7S1 A0A0C5D7U2 A0A0C5DCE7 A0A0C5DCF9 A0A0C5DGJ4 A0A0C5DUB7 A0A0C5DGG7 A0A0C5DGL8 A0A2A4J6K8 A0A2H1WIH9 A0A0C5DU51 A0A1E1W7P6 A0A0C5DU79 G3K8G5 G3K8E0 G3K8D7 A0A0C5D7P6 A0A0C5D7S8 G3K8E2 G3K8G8 G3K8E7 G3K8G4 A0A0C5DGH1 A0A3S2NYT8 A0A0C5DBR1 A0A0C5D7R3 A0A0C5DGH9 A0A0C5DCC3 A0A0C5DU59 A0A0C5DCI8 A0A0C5DCI4
EMBL
BABH01013510
DQ311242
ABD36187.1
KQ460968
KPJ10226.1
KQ459324
+ More
KPJ01569.1 KP074185 AJO66019.1 KP074194 AJO66028.1 KP074223 AJO66057.1 KP074235 AJO66069.1 KP074236 AJO66070.1 KP074278 AJO66112.1 KP074237 AJO66071.1 KP074281 AJO66115.1 KP074248 AJO66082.1 KP074217 AJO66051.1 KP074207 AJO66041.1 KP074228 AJO66062.1 KP074227 AJO66061.1 KP074230 AJO66064.1 KP074231 AJO66065.1 KP074229 AJO66063.1 KP074234 AJO66068.1 KP074233 AJO66067.1 KP074211 AJO66045.1 KP074209 KP074210 AJO66044.1 KP074283 KP074284 AJO66118.1 KP074279 KP074282 AJO66116.1 KP074196 AJO66030.1 KP074195 AJO66029.1 KP074197 AJO66031.1 KP074298 AJO66132.1 KP074198 AJO66032.1 KP074189 KP074190 AJO66023.1 KP074297 AJO66131.1 KP074287 AJO66121.1 KP074205 AJO66039.1 KP074272 AJO66106.1 KP074243 AJO66077.1 KP074204 AJO66038.1 KP074271 AJO66105.1 KP074206 AJO66040.1 KP074257 AJO66091.1 KP074259 AJO66093.1 KP074267 AJO66101.1 KP074200 AJO66034.1 KP074263 AJO66097.1 KP074246 AJO66080.1 KP074285 AJO66119.1 KP074256 AJO66090.1 KP074258 AJO66092.1 KP074270 AJO66104.1 KP074262 AJO66096.1 KP074291 KP074292 AJO66125.1 KP074289 KP074290 AJO66124.1 KP074249 KP074250 KP074251 AJO66084.1 KP074264 KP074265 AJO66098.1 KP074213 KP074214 AJO66048.1 KP074242 KP074245 AJO66076.1 KP074238 KP074239 AJO66072.1 KP074286 KP074288 AJO66120.1 KP074215 KP074216 AJO66050.1 KP074260 KP074261 AJO66095.1 KP074273 KP074274 AJO66108.1 KP074225 KP074226 AJO66060.1 GAIX01003343 JAA89217.1 JTDY01001172 KOB74659.1 KP074183 AJO66017.1 KP074186 AJO66020.1 KP074294 AJO66128.1 KP074295 AJO66129.1 KP074247 AJO66081.1 KP074222 AJO66056.1 KP074252 KP074253 AJO66086.1 KP074218 KP074219 KP074220 AJO66054.1 KP074240 AJO66074.1 KP074224 AJO66058.1 KP074276 AJO66110.1 KP074179 AJO66013.1 KP074269 AJO66103.1 NWSH01003030 PCG67080.1 ODYU01008823 SOQ52766.1 KP074181 AJO66015.1 GDQN01008075 GDQN01005056 JAT82979.1 JAT85998.1 KP074221 AJO66055.1 JN174280 JN174281 JN174282 JN174283 JN174284 JN174285 JN174287 JN174288 JN174289 JN174291 JN174292 JN174293 JN174294 AEO01858.1 AEO01859.1 AEO01860.1 AEO01861.1 AEO01862.1 AEO01863.1 AEO01865.1 AEO01866.1 AEO01867.1 AEO01869.1 AEO01870.1 AEO01871.1 AEO01872.1 JN174261 JN174262 JN174263 JN174265 JN174266 JN174267 JN174268 JN174270 JN174271 JN174272 JN174273 JN174274 JN174275 JN174276 JN174277 JN174278 JN174279 AEO01839.1 AEO01840.1 AEO01841.1 AEO01843.1 AEO01844.1 AEO01845.1 AEO01846.1 AEO01848.1 AEO01849.1 AEO01850.1 AEO01851.1 AEO01852.1 AEO01853.1 AEO01854.1 AEO01855.1 AEO01856.1 AEO01857.1 JN174259 JN174260 AEO01837.1 AEO01838.1 KP074187 AJO66021.1 KP074232 AJO66066.1 JN174264 AEO01842.1 JN174290 AEO01868.1 JN174269 AEO01847.1 JN174286 AEO01864.1 KP074184 AJO66018.1 RSAL01000021 RVE52506.1 KP074193 AJO66027.1 KP074212 AJO66046.1 KP074199 AJO66033.1 KP074180 AJO66014.1 KP074191 AJO66025.1 KP074280 AJO66114.1 KP074275 AJO66109.1
KPJ01569.1 KP074185 AJO66019.1 KP074194 AJO66028.1 KP074223 AJO66057.1 KP074235 AJO66069.1 KP074236 AJO66070.1 KP074278 AJO66112.1 KP074237 AJO66071.1 KP074281 AJO66115.1 KP074248 AJO66082.1 KP074217 AJO66051.1 KP074207 AJO66041.1 KP074228 AJO66062.1 KP074227 AJO66061.1 KP074230 AJO66064.1 KP074231 AJO66065.1 KP074229 AJO66063.1 KP074234 AJO66068.1 KP074233 AJO66067.1 KP074211 AJO66045.1 KP074209 KP074210 AJO66044.1 KP074283 KP074284 AJO66118.1 KP074279 KP074282 AJO66116.1 KP074196 AJO66030.1 KP074195 AJO66029.1 KP074197 AJO66031.1 KP074298 AJO66132.1 KP074198 AJO66032.1 KP074189 KP074190 AJO66023.1 KP074297 AJO66131.1 KP074287 AJO66121.1 KP074205 AJO66039.1 KP074272 AJO66106.1 KP074243 AJO66077.1 KP074204 AJO66038.1 KP074271 AJO66105.1 KP074206 AJO66040.1 KP074257 AJO66091.1 KP074259 AJO66093.1 KP074267 AJO66101.1 KP074200 AJO66034.1 KP074263 AJO66097.1 KP074246 AJO66080.1 KP074285 AJO66119.1 KP074256 AJO66090.1 KP074258 AJO66092.1 KP074270 AJO66104.1 KP074262 AJO66096.1 KP074291 KP074292 AJO66125.1 KP074289 KP074290 AJO66124.1 KP074249 KP074250 KP074251 AJO66084.1 KP074264 KP074265 AJO66098.1 KP074213 KP074214 AJO66048.1 KP074242 KP074245 AJO66076.1 KP074238 KP074239 AJO66072.1 KP074286 KP074288 AJO66120.1 KP074215 KP074216 AJO66050.1 KP074260 KP074261 AJO66095.1 KP074273 KP074274 AJO66108.1 KP074225 KP074226 AJO66060.1 GAIX01003343 JAA89217.1 JTDY01001172 KOB74659.1 KP074183 AJO66017.1 KP074186 AJO66020.1 KP074294 AJO66128.1 KP074295 AJO66129.1 KP074247 AJO66081.1 KP074222 AJO66056.1 KP074252 KP074253 AJO66086.1 KP074218 KP074219 KP074220 AJO66054.1 KP074240 AJO66074.1 KP074224 AJO66058.1 KP074276 AJO66110.1 KP074179 AJO66013.1 KP074269 AJO66103.1 NWSH01003030 PCG67080.1 ODYU01008823 SOQ52766.1 KP074181 AJO66015.1 GDQN01008075 GDQN01005056 JAT82979.1 JAT85998.1 KP074221 AJO66055.1 JN174280 JN174281 JN174282 JN174283 JN174284 JN174285 JN174287 JN174288 JN174289 JN174291 JN174292 JN174293 JN174294 AEO01858.1 AEO01859.1 AEO01860.1 AEO01861.1 AEO01862.1 AEO01863.1 AEO01865.1 AEO01866.1 AEO01867.1 AEO01869.1 AEO01870.1 AEO01871.1 AEO01872.1 JN174261 JN174262 JN174263 JN174265 JN174266 JN174267 JN174268 JN174270 JN174271 JN174272 JN174273 JN174274 JN174275 JN174276 JN174277 JN174278 JN174279 AEO01839.1 AEO01840.1 AEO01841.1 AEO01843.1 AEO01844.1 AEO01845.1 AEO01846.1 AEO01848.1 AEO01849.1 AEO01850.1 AEO01851.1 AEO01852.1 AEO01853.1 AEO01854.1 AEO01855.1 AEO01856.1 AEO01857.1 JN174259 JN174260 AEO01837.1 AEO01838.1 KP074187 AJO66021.1 KP074232 AJO66066.1 JN174264 AEO01842.1 JN174290 AEO01868.1 JN174269 AEO01847.1 JN174286 AEO01864.1 KP074184 AJO66018.1 RSAL01000021 RVE52506.1 KP074193 AJO66027.1 KP074212 AJO66046.1 KP074199 AJO66033.1 KP074180 AJO66014.1 KP074191 AJO66025.1 KP074280 AJO66114.1 KP074275 AJO66109.1
Proteomes
PRIDE
Pfam
PF00137 ATP-synt_C
Interpro
SUPFAM
SSF81333
SSF81333
ProteinModelPortal
Q2F630
A0A0N0PBL6
A0A194Q852
A0A0C5DCC5
A0A0C5DGH6
A0A0C5DBS7
+ More
A0A0C5DCF6 A0A0C5DU88 A0A0C5DBW0 A0A0C5D7T1 A0A0C5DUC0 A0A0C5DBU1 A0A0C5D7R7 A0A0C5D7R1 A0A0C5DBT1 A0A0C5D7S5 A0A0C5DCF3 A0A0C5DU86 A0A0C5DGJ6 A0A0C5DGJ7 A0A0C5DBT3 A0A0C5DU73 A0A0C5DCE0 A0A0C5DGM6 A0A0C5D7W4 A0A0C5DU61 A0A0C5DCD2 A0A0C5D7Q3 A0A0C5DBX3 A0A0C5DBR5 A0A0C5DGH5 A0A0C5D7X7 A0A0C5D7W9 A0A0C5DCD8 A0A0C5D7V7 A0A0C5DBT9 A0A0C5DGI3 A0A0C5DUB3 A0A0C5DU68 A0A0C5D7U6 A0A0C5DGL3 A0A0C5D7V3 A0A0C5DCD4 A0A0C5DBV1 A0A0C5DU94 A0A0C5DCJ3 A0A0C5DUA2 A0A0C5DBU8 A0A0C5DCI0 A0A0C5D7U8 A0A0C5DUD0 A0A0C5DCJ7 A0A0C5DCG5 A0A0C5DGL5 A0A0C5DGI9 A0A0C5D7T6 A0A0C5DBT6 A0A0C5DUC5 A0A0C5DU76 A0A0C5DUA6 A0A0C5DGM0 A0A0C5DU83 S4PYG2 A0A0L7LH25 A0A0C5DBQ7 A0A0C5DU55 A0A0C5DGN3 A0A0C5DCK2 A0A0C5D7T9 A0A0C5D7S1 A0A0C5D7U2 A0A0C5DCE7 A0A0C5DCF9 A0A0C5DGJ4 A0A0C5DUB7 A0A0C5DGG7 A0A0C5DGL8 A0A2A4J6K8 A0A2H1WIH9 A0A0C5DU51 A0A1E1W7P6 A0A0C5DU79 G3K8G5 G3K8E0 G3K8D7 A0A0C5D7P6 A0A0C5D7S8 G3K8E2 G3K8G8 G3K8E7 G3K8G4 A0A0C5DGH1 A0A3S2NYT8 A0A0C5DBR1 A0A0C5D7R3 A0A0C5DGH9 A0A0C5DCC3 A0A0C5DU59 A0A0C5DCI8 A0A0C5DCI4
A0A0C5DCF6 A0A0C5DU88 A0A0C5DBW0 A0A0C5D7T1 A0A0C5DUC0 A0A0C5DBU1 A0A0C5D7R7 A0A0C5D7R1 A0A0C5DBT1 A0A0C5D7S5 A0A0C5DCF3 A0A0C5DU86 A0A0C5DGJ6 A0A0C5DGJ7 A0A0C5DBT3 A0A0C5DU73 A0A0C5DCE0 A0A0C5DGM6 A0A0C5D7W4 A0A0C5DU61 A0A0C5DCD2 A0A0C5D7Q3 A0A0C5DBX3 A0A0C5DBR5 A0A0C5DGH5 A0A0C5D7X7 A0A0C5D7W9 A0A0C5DCD8 A0A0C5D7V7 A0A0C5DBT9 A0A0C5DGI3 A0A0C5DUB3 A0A0C5DU68 A0A0C5D7U6 A0A0C5DGL3 A0A0C5D7V3 A0A0C5DCD4 A0A0C5DBV1 A0A0C5DU94 A0A0C5DCJ3 A0A0C5DUA2 A0A0C5DBU8 A0A0C5DCI0 A0A0C5D7U8 A0A0C5DUD0 A0A0C5DCJ7 A0A0C5DCG5 A0A0C5DGL5 A0A0C5DGI9 A0A0C5D7T6 A0A0C5DBT6 A0A0C5DUC5 A0A0C5DU76 A0A0C5DUA6 A0A0C5DGM0 A0A0C5DU83 S4PYG2 A0A0L7LH25 A0A0C5DBQ7 A0A0C5DU55 A0A0C5DGN3 A0A0C5DCK2 A0A0C5D7T9 A0A0C5D7S1 A0A0C5D7U2 A0A0C5DCE7 A0A0C5DCF9 A0A0C5DGJ4 A0A0C5DUB7 A0A0C5DGG7 A0A0C5DGL8 A0A2A4J6K8 A0A2H1WIH9 A0A0C5DU51 A0A1E1W7P6 A0A0C5DU79 G3K8G5 G3K8E0 G3K8D7 A0A0C5D7P6 A0A0C5D7S8 G3K8E2 G3K8G8 G3K8E7 G3K8G4 A0A0C5DGH1 A0A3S2NYT8 A0A0C5DBR1 A0A0C5D7R3 A0A0C5DGH9 A0A0C5DCC3 A0A0C5DU59 A0A0C5DCI8 A0A0C5DCI4
PDB
6O7X
E-value=1.71748e-30,
Score=327
Ontologies
PATHWAY
GO
Topology
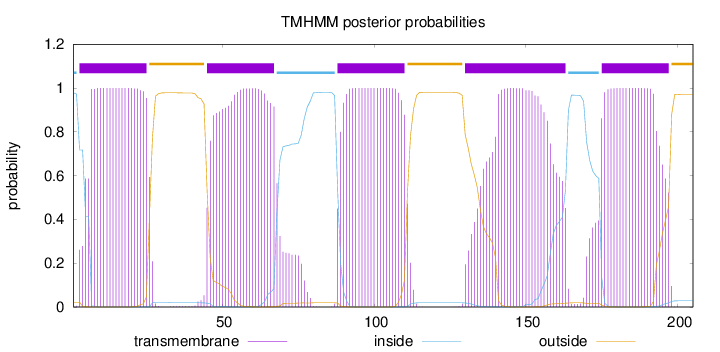
Length:
205
Number of predicted TMHs:
5
Exp number of AAs in TMHs:
117.1575
Exp number, first 60 AAs:
36.0591
Total prob of N-in:
0.97812
POSSIBLE N-term signal
sequence
inside
1 - 2
TMhelix
3 - 25
outside
26 - 44
TMhelix
45 - 67
inside
68 - 87
TMhelix
88 - 110
outside
111 - 129
TMhelix
130 - 163
inside
164 - 174
TMhelix
175 - 197
outside
198 - 205
Population Genetic Test Statistics
Pi
197.217926
Theta
184.451069
Tajima's D
-1.188017
CLR
165.088283
CSRT
0.106394680265987
Interpretation
Uncertain
