Gene
KWMTBOMO12677
Annotation
endonuclease-reverse_transcriptase_[Bombyx_mori]
Location in the cell
Nuclear Reliability : 3.343
Sequence
CDS
ATGGTAGTTTCAAGAAACCCGGATTTGAGTTCAAGCGTATCTGTAGCTGGGAAACAACTAGAGCGGGTACGACAGTACAAGTATTTAGGAGCTTGGGTAAATGAAGCATGGGAATCTGATCAAGAAATCAAGACCCGGATAGAAACTGCTCAAGCCTCTTTTAATAAAATGAGGAAAGTTCTTTGCTGTCTTCAACTGAATATAAAGCTCCGAGTCAGGCTACTCATGTGCTATATCTGGCCTATCGTCATGTATGGTTGCGAAACGTGGACCCTGAAAGAGGACACTACAAGACGCCTACAGGCATTCGAGATGTGGTGCTACCGTCGCATGTTACGCATTAGTTGGACGCAGAGGGTCAGGAACGAAACCGTGCTTCAGCGCGTTCATATGTCCCGGAAAATGTTGCCTTCTATCAAAAAGCGCAAGACAGAGTATCTCGGCCACGTGCTCAGGAACGATCGATACATGCTGCTGCAGCTGATAATTATGGGCAAAATGGACGGAAAGAGACGTGCTGGGCGAAGAAAAAAGTCATGGCTCCGGAATATCCGGGAGTGGACCGGTATTGCATCCGTCGAACAGCTGTTCCAGCTGGCTGCTGACAGAAAAGAATACAGGAAGCTAACGGCCAACCTTCAGGATTGA
Protein
MVVSRNPDLSSSVSVAGKQLERVRQYKYLGAWVNEAWESDQEIKTRIETAQASFNKMRKVLCCLQLNIKLRVRLLMCYIWPIVMYGCETWTLKEDTTRRLQAFEMWCYRRMLRISWTQRVRNETVLQRVHMSRKMLPSIKKRKTEYLGHVLRNDRYMLLQLIIMGKMDGKRRAGRRKKSWLRNIREWTGIASVEQLFQLAADRKEYRKLTANLQD
Summary
Uniprot
D5LB38
A0A2A4J795
A0A146L0Z1
A0A0A9XCF5
A0A0A9XXL9
A0A0J7KRQ5
+ More
A0A0J7KZE8 A0A2S2QAF2 A0A2A4JBK9 S4NFP7 A0A0K8T3U8 A0A2S2NMQ2 A0A3Q0JFR3 A0A0B7BSZ9 A0A0B7BUR9 A0A0B7BT80 A0A0B7BVD9 A0A0B7BVD3 A0A0B7BSB6 E2C8Z8 A0A2J7RG81 A0A2J7R045 A0A2J7QN33 A0A2J7PGF8 A0A2J7Q676 A0A2J7Q7P8 A0A2J7PZP5 A0A2S2PZ46 A0A2J7PDC6 A0A2J7Q4D3 A0A2J7RTL3 A0A2J7QV96 A0A2J7RNL1 A0A2J7RPC0 A0A2J7PNF2 A0A2J7RQ44 A0A2J7RQG9 A0A2J7RMV2 A0A2J7QAH8 A0A2J7PWM3 A0A2S2PJ33 X1XQS0 A0A2J7QXG8 A0A2J7QIG5 A0A2J7QAN0 A0A2J7QEA2 A0A2J7QUT4 A0A2J7RH22 A0A2J7PF56 A0A2J7QZ54 A0A2J7RKH7 A0A2J7PVJ3 A0A2J7RLP3 A0A2J7RSJ5 A0A2J7QJ13 A0A2J7QEN3 A0A2J7Q6A8 A0A2J7QR14 A0A2J7PWU5 A0A2J7QVI2 A0A2J7PWV5 A0A2J7QSD2 A0A2J7RS69 A0A2J7PF33 A0A2J7PDY4 A0A2J7R4G7 A0A2J7RFE8 J9KDM4 A0A2J7QME3 A0A2J7PXJ3 A0A2J7R4L3 A0A2J7R3G6 A0A2J7PMA7 A0A2J7QVS6 A0A2J7Q420 A0A2J7QLG4 A0A2J7PFP6 A0A2J7R076 A0A2J7Q0H2 A0A2J7RMV7 A0A2J7RAR3 A0A2J7PJ38 A0A2J7RDL7 A0A2J7RM57 A0A2J7QD50 A0A2J7QP57 A0A2J7PJ03 A0A2J7RLC6 A0A2J7QR00 A0A2J7Q7C4 A0A2J7Q5D5 A0A2S2Q0B1 A0A2J7RIN3 A0A2J7PGM6 A0A2J7PBF1 A0A2J7RKX8
A0A0J7KZE8 A0A2S2QAF2 A0A2A4JBK9 S4NFP7 A0A0K8T3U8 A0A2S2NMQ2 A0A3Q0JFR3 A0A0B7BSZ9 A0A0B7BUR9 A0A0B7BT80 A0A0B7BVD9 A0A0B7BVD3 A0A0B7BSB6 E2C8Z8 A0A2J7RG81 A0A2J7R045 A0A2J7QN33 A0A2J7PGF8 A0A2J7Q676 A0A2J7Q7P8 A0A2J7PZP5 A0A2S2PZ46 A0A2J7PDC6 A0A2J7Q4D3 A0A2J7RTL3 A0A2J7QV96 A0A2J7RNL1 A0A2J7RPC0 A0A2J7PNF2 A0A2J7RQ44 A0A2J7RQG9 A0A2J7RMV2 A0A2J7QAH8 A0A2J7PWM3 A0A2S2PJ33 X1XQS0 A0A2J7QXG8 A0A2J7QIG5 A0A2J7QAN0 A0A2J7QEA2 A0A2J7QUT4 A0A2J7RH22 A0A2J7PF56 A0A2J7QZ54 A0A2J7RKH7 A0A2J7PVJ3 A0A2J7RLP3 A0A2J7RSJ5 A0A2J7QJ13 A0A2J7QEN3 A0A2J7Q6A8 A0A2J7QR14 A0A2J7PWU5 A0A2J7QVI2 A0A2J7PWV5 A0A2J7QSD2 A0A2J7RS69 A0A2J7PF33 A0A2J7PDY4 A0A2J7R4G7 A0A2J7RFE8 J9KDM4 A0A2J7QME3 A0A2J7PXJ3 A0A2J7R4L3 A0A2J7R3G6 A0A2J7PMA7 A0A2J7QVS6 A0A2J7Q420 A0A2J7QLG4 A0A2J7PFP6 A0A2J7R076 A0A2J7Q0H2 A0A2J7RMV7 A0A2J7RAR3 A0A2J7PJ38 A0A2J7RDL7 A0A2J7RM57 A0A2J7QD50 A0A2J7QP57 A0A2J7PJ03 A0A2J7RLC6 A0A2J7QR00 A0A2J7Q7C4 A0A2J7Q5D5 A0A2S2Q0B1 A0A2J7RIN3 A0A2J7PGM6 A0A2J7PBF1 A0A2J7RKX8
EMBL
GU815089
ADF18552.1
NWSH01002736
PCG67606.1
GDHC01017214
JAQ01415.1
+ More
GBHO01028834 JAG14770.1 GBHO01020011 GDHC01013600 JAG23593.1 JAQ05029.1 LBMM01003995 KMQ92919.1 LBMM01001656 KMQ95932.1 GGMS01004979 MBY74182.1 NWSH01001998 PCG69477.1 GAIX01014959 JAA77601.1 GBRD01005612 JAG60209.1 GGMR01005851 MBY18470.1 HACG01049248 HACG01049253 CEK96113.1 CEK96118.1 HACG01049251 HACG01049252 CEK96116.1 CEK96117.1 HACG01049244 HACG01049247 CEK96109.1 CEK96112.1 HACG01049255 CEK96120.1 HACG01049245 CEK96110.1 HACG01049249 CEK96114.1 GL453752 EFN75584.1 NEVH01004403 PNF39833.1 NEVH01008300 PNF34184.1 NEVH01013201 PNF29989.1 NEVH01025635 PNF15414.1 NEVH01017535 PNF24084.1 NEVH01017441 PNF24607.1 NEVH01020333 PNF21801.1 GGMS01001437 MBY70640.1 NEVH01026391 PNF14322.1 NEVH01018384 PNF23448.1 NEVH01000001 PNF44158.1 NEVH01010478 PNF32508.1 NEVH01002157 PNF42414.1 NEVH01001374 PNF42669.1 NEVH01023953 PNF17874.1 NEVH01001347 PNF42965.1 NEVH01001337 PNF43070.1 NEVH01002549 PNF42160.1 NEVH01016330 PNF25584.1 NEVH01020870 PNF20747.1 GGMR01016842 MBY29461.1 ABLF02038495 NEVH01009381 PNF33254.1 NEVH01013652 PNF28353.1 NEVH01016329 PNF25648.1 NEVH01015318 PNF26901.1 NEVH01010487 PNF32335.1 NEVH01003750 PNF40097.1 NEVH01026085 PNF14958.1 NEVH01009076 PNF33845.1 NEVH01002728 PNF41343.1 NEVH01020956 NEVH01016294 NEVH01011197 NEVH01009765 NEVH01003008 PNF20357.1 PNF26083.1 PNF31821.1 PNF32947.1 PNF40845.1 NEVH01002684 PNF41750.1 NEVH01000257 PNF43803.1 NEVH01013563 PNF28562.1 NEVH01015308 PNF27027.1 NEVH01017534 PNF24114.1 NEVH01012081 PNF31013.1 NEVH01020868 PNF20801.1 NEVH01009907 PNF32598.1 NEVH01020867 PNF20816.1 NEVH01011876 PNF31501.1 NEVH01000295 PNF43662.1 PNF14932.1 NEVH01026122 PNF14543.1 NEVH01007419 PNF35717.1 NEVH01004413 PNF39563.1 ABLF02046942 ABLF02059971 NEVH01013210 PNF29764.1 NEVH01020856 PNF21059.1 NEVH01007405 PNF35773.1 NEVH01007819 PNF35377.1 NEVH01024189 PNF17475.1 NEVH01009772 PNF32674.1 NEVH01018411 PNF23333.1 NEVH01013245 PNF29434.1 NEVH01025651 PNF15155.1 NEVH01008283 PNF34242.1 NEVH01019967 PNF22088.1 PNF42173.1 NEVH01006565 PNF37921.1 NEVH01024952 PNF16338.1 NEVH01005288 PNF38924.1 NEVH01002574 PNF41923.1 NEVH01015827 PNF26505.1 NEVH01012097 PNF30370.1 PNF16321.1 NEVH01002690 PNF41636.1 PNF31014.1 NEVH01017443 PNF24479.1 NEVH01017570 PNF23798.1 GGMS01001369 MBY70572.1 NEVH01003494 PNF40693.1 NEVH01025174 PNF15486.1 NEVH01027104 PNF13657.1 NEVH01002704 PNF41497.1
GBHO01028834 JAG14770.1 GBHO01020011 GDHC01013600 JAG23593.1 JAQ05029.1 LBMM01003995 KMQ92919.1 LBMM01001656 KMQ95932.1 GGMS01004979 MBY74182.1 NWSH01001998 PCG69477.1 GAIX01014959 JAA77601.1 GBRD01005612 JAG60209.1 GGMR01005851 MBY18470.1 HACG01049248 HACG01049253 CEK96113.1 CEK96118.1 HACG01049251 HACG01049252 CEK96116.1 CEK96117.1 HACG01049244 HACG01049247 CEK96109.1 CEK96112.1 HACG01049255 CEK96120.1 HACG01049245 CEK96110.1 HACG01049249 CEK96114.1 GL453752 EFN75584.1 NEVH01004403 PNF39833.1 NEVH01008300 PNF34184.1 NEVH01013201 PNF29989.1 NEVH01025635 PNF15414.1 NEVH01017535 PNF24084.1 NEVH01017441 PNF24607.1 NEVH01020333 PNF21801.1 GGMS01001437 MBY70640.1 NEVH01026391 PNF14322.1 NEVH01018384 PNF23448.1 NEVH01000001 PNF44158.1 NEVH01010478 PNF32508.1 NEVH01002157 PNF42414.1 NEVH01001374 PNF42669.1 NEVH01023953 PNF17874.1 NEVH01001347 PNF42965.1 NEVH01001337 PNF43070.1 NEVH01002549 PNF42160.1 NEVH01016330 PNF25584.1 NEVH01020870 PNF20747.1 GGMR01016842 MBY29461.1 ABLF02038495 NEVH01009381 PNF33254.1 NEVH01013652 PNF28353.1 NEVH01016329 PNF25648.1 NEVH01015318 PNF26901.1 NEVH01010487 PNF32335.1 NEVH01003750 PNF40097.1 NEVH01026085 PNF14958.1 NEVH01009076 PNF33845.1 NEVH01002728 PNF41343.1 NEVH01020956 NEVH01016294 NEVH01011197 NEVH01009765 NEVH01003008 PNF20357.1 PNF26083.1 PNF31821.1 PNF32947.1 PNF40845.1 NEVH01002684 PNF41750.1 NEVH01000257 PNF43803.1 NEVH01013563 PNF28562.1 NEVH01015308 PNF27027.1 NEVH01017534 PNF24114.1 NEVH01012081 PNF31013.1 NEVH01020868 PNF20801.1 NEVH01009907 PNF32598.1 NEVH01020867 PNF20816.1 NEVH01011876 PNF31501.1 NEVH01000295 PNF43662.1 PNF14932.1 NEVH01026122 PNF14543.1 NEVH01007419 PNF35717.1 NEVH01004413 PNF39563.1 ABLF02046942 ABLF02059971 NEVH01013210 PNF29764.1 NEVH01020856 PNF21059.1 NEVH01007405 PNF35773.1 NEVH01007819 PNF35377.1 NEVH01024189 PNF17475.1 NEVH01009772 PNF32674.1 NEVH01018411 PNF23333.1 NEVH01013245 PNF29434.1 NEVH01025651 PNF15155.1 NEVH01008283 PNF34242.1 NEVH01019967 PNF22088.1 PNF42173.1 NEVH01006565 PNF37921.1 NEVH01024952 PNF16338.1 NEVH01005288 PNF38924.1 NEVH01002574 PNF41923.1 NEVH01015827 PNF26505.1 NEVH01012097 PNF30370.1 PNF16321.1 NEVH01002690 PNF41636.1 PNF31014.1 NEVH01017443 PNF24479.1 NEVH01017570 PNF23798.1 GGMS01001369 MBY70572.1 NEVH01003494 PNF40693.1 NEVH01025174 PNF15486.1 NEVH01027104 PNF13657.1 NEVH01002704 PNF41497.1
Proteomes
PRIDE
Interpro
SUPFAM
SSF56219
SSF56219
Gene 3D
ProteinModelPortal
D5LB38
A0A2A4J795
A0A146L0Z1
A0A0A9XCF5
A0A0A9XXL9
A0A0J7KRQ5
+ More
A0A0J7KZE8 A0A2S2QAF2 A0A2A4JBK9 S4NFP7 A0A0K8T3U8 A0A2S2NMQ2 A0A3Q0JFR3 A0A0B7BSZ9 A0A0B7BUR9 A0A0B7BT80 A0A0B7BVD9 A0A0B7BVD3 A0A0B7BSB6 E2C8Z8 A0A2J7RG81 A0A2J7R045 A0A2J7QN33 A0A2J7PGF8 A0A2J7Q676 A0A2J7Q7P8 A0A2J7PZP5 A0A2S2PZ46 A0A2J7PDC6 A0A2J7Q4D3 A0A2J7RTL3 A0A2J7QV96 A0A2J7RNL1 A0A2J7RPC0 A0A2J7PNF2 A0A2J7RQ44 A0A2J7RQG9 A0A2J7RMV2 A0A2J7QAH8 A0A2J7PWM3 A0A2S2PJ33 X1XQS0 A0A2J7QXG8 A0A2J7QIG5 A0A2J7QAN0 A0A2J7QEA2 A0A2J7QUT4 A0A2J7RH22 A0A2J7PF56 A0A2J7QZ54 A0A2J7RKH7 A0A2J7PVJ3 A0A2J7RLP3 A0A2J7RSJ5 A0A2J7QJ13 A0A2J7QEN3 A0A2J7Q6A8 A0A2J7QR14 A0A2J7PWU5 A0A2J7QVI2 A0A2J7PWV5 A0A2J7QSD2 A0A2J7RS69 A0A2J7PF33 A0A2J7PDY4 A0A2J7R4G7 A0A2J7RFE8 J9KDM4 A0A2J7QME3 A0A2J7PXJ3 A0A2J7R4L3 A0A2J7R3G6 A0A2J7PMA7 A0A2J7QVS6 A0A2J7Q420 A0A2J7QLG4 A0A2J7PFP6 A0A2J7R076 A0A2J7Q0H2 A0A2J7RMV7 A0A2J7RAR3 A0A2J7PJ38 A0A2J7RDL7 A0A2J7RM57 A0A2J7QD50 A0A2J7QP57 A0A2J7PJ03 A0A2J7RLC6 A0A2J7QR00 A0A2J7Q7C4 A0A2J7Q5D5 A0A2S2Q0B1 A0A2J7RIN3 A0A2J7PGM6 A0A2J7PBF1 A0A2J7RKX8
A0A0J7KZE8 A0A2S2QAF2 A0A2A4JBK9 S4NFP7 A0A0K8T3U8 A0A2S2NMQ2 A0A3Q0JFR3 A0A0B7BSZ9 A0A0B7BUR9 A0A0B7BT80 A0A0B7BVD9 A0A0B7BVD3 A0A0B7BSB6 E2C8Z8 A0A2J7RG81 A0A2J7R045 A0A2J7QN33 A0A2J7PGF8 A0A2J7Q676 A0A2J7Q7P8 A0A2J7PZP5 A0A2S2PZ46 A0A2J7PDC6 A0A2J7Q4D3 A0A2J7RTL3 A0A2J7QV96 A0A2J7RNL1 A0A2J7RPC0 A0A2J7PNF2 A0A2J7RQ44 A0A2J7RQG9 A0A2J7RMV2 A0A2J7QAH8 A0A2J7PWM3 A0A2S2PJ33 X1XQS0 A0A2J7QXG8 A0A2J7QIG5 A0A2J7QAN0 A0A2J7QEA2 A0A2J7QUT4 A0A2J7RH22 A0A2J7PF56 A0A2J7QZ54 A0A2J7RKH7 A0A2J7PVJ3 A0A2J7RLP3 A0A2J7RSJ5 A0A2J7QJ13 A0A2J7QEN3 A0A2J7Q6A8 A0A2J7QR14 A0A2J7PWU5 A0A2J7QVI2 A0A2J7PWV5 A0A2J7QSD2 A0A2J7RS69 A0A2J7PF33 A0A2J7PDY4 A0A2J7R4G7 A0A2J7RFE8 J9KDM4 A0A2J7QME3 A0A2J7PXJ3 A0A2J7R4L3 A0A2J7R3G6 A0A2J7PMA7 A0A2J7QVS6 A0A2J7Q420 A0A2J7QLG4 A0A2J7PFP6 A0A2J7R076 A0A2J7Q0H2 A0A2J7RMV7 A0A2J7RAR3 A0A2J7PJ38 A0A2J7RDL7 A0A2J7RM57 A0A2J7QD50 A0A2J7QP57 A0A2J7PJ03 A0A2J7RLC6 A0A2J7QR00 A0A2J7Q7C4 A0A2J7Q5D5 A0A2S2Q0B1 A0A2J7RIN3 A0A2J7PGM6 A0A2J7PBF1 A0A2J7RKX8
Ontologies
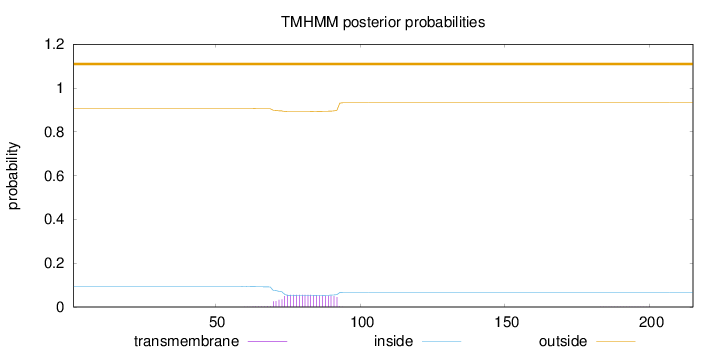
Topology
Length:
215
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
1.16018
Exp number, first 60 AAs:
0.00062
Total prob of N-in:
0.09419
outside
1 - 215
Population Genetic Test Statistics
Pi
2.775001
Theta
7.951037
Tajima's D
-1.990642
CLR
184.929872
CSRT
0.0116994150292485
Interpretation
Uncertain
