Gene
KWMTBOMO09728
Pre Gene Modal
BGIBMGA012892
Annotation
PREDICTED:_luciferin_4-monooxygenase-like_isoform_X1_[Bombyx_mori]
Full name
Luciferin 4-monooxygenase
Location in the cell
Cytoplasmic Reliability : 1.522 Mitochondrial Reliability : 1.272 Nuclear Reliability : 1.486
Sequence
CDS
ATGTACTGGGATGAGCACTTTTATTTTTACTTTGTAGAGCGACTAAAGCTGTTGATTCATTACAAGAATCATCAGATATCACCGCTTGAAATAGAGAAGGTCATTCGCCAGCATCCGGGTGTGTTGGACGTTATAGTCACCAGCATCACGGACGTCCAGCGCGAGGAGCTGCCGTGCGCTTGTGTCGTGCTTAAAGATGGACGTCGCGTCACCGAACAGGAAATCAAGGACTTGGTTCGAGATTCGCTAACGGATCCAAAACAATTGAGAGGTGGCGTTTTTTTCTTAAAGGAAATGCCCATGAACCCACAACTGAAAATCGACAGAAGAAAAATAAAAGAATTCGTTATTAATTCATTAGGCACAAAAAAATAA
Protein
MYWDEHFYFYFVERLKLLIHYKNHQISPLEIEKVIRQHPGVLDVIVTSITDVQREELPCACVVLKDGRRVTEQEIKDLVRDSLTDPKQLRGGVFFLKEMPMNPQLKIDRRKIKEFVINSLGTKK
Summary
Description
Produces green light with a wavelength of 562 nm.
Catalytic Activity
ATP + firefly D-luciferin + O2 = AMP + CO2 + diphosphate + firefly oxyluciferin + hnu
Cofactor
Mg(2+)
Similarity
Belongs to the ATP-dependent AMP-binding enzyme family.
Belongs to the peptidase S1 family.
Belongs to the peptidase S1 family.
Keywords
3D-structure
ATP-binding
Luminescence
Magnesium
Metal-binding
Monooxygenase
Nucleotide-binding
Oxidoreductase
Peroxisome
Photoprotein
Feature
chain Luciferin 4-monooxygenase
Uniprot
A0A0N1IQK8
A0A194PZY0
A0A194R6T5
A0A1B3TP04
A0A1B3TNX4
A0A1B3TNY4
+ More
A0A2H1VFT6 A0A2W1C0Z5 A0A2A4JE16 A0A0L7LS68 A0A1B3TNZ3 A0A1B3TP14 A0A2W1BFJ2 A0A2A4JFU1 A0A1B3TNX9 A0A1B3TP12 A0A2A4JCF3 A0A1B3TNZ2 A0A1B3TNX2 A0A1B3TP25 A0A2W1BJK0 A0A1B3TP11 A0A1B3TP22 A0A3S2N8I8 A0A2H1V3V7 A0A1B3TNZ6 A0A2W1BLY3 A0A2A4J5M1 A0A194Q640 A0A1B3TP10 A0A1B3TNZ4 A0A212FBS4 A0A2H1VAR1 A0A2A4JDK9 A0A1B3TP07 A0A2H1VV91 A0A2A4JGT9 A0A2W1BJT9 A0A2H1WV86 A0A1B3TNX3 A0A2H1W204 A0A1B3TNY0 A0A194Q0P0 A0A1B3TNZ0 A0A0N1PKC2 A0A2A4J4G8 A0A1B3TNZ5 A0A194PZW6 A0A2W1BGF2 A0A1B3TNW9 A0A1B3TP01 A0A212FN15 A0A194Q1K5 A0A194R1K6 A0A194R232 A0A1B3TP24 A0A2H1V5T4 A0A2H1V0G3 A0A2H1V5V8 A0A2H1VH10 A0A2H1WMC6 A0A2T6WK65 A0A2A4JKH2 A0A2W1B861 A0A428D0L8 A0A2S3QZT6 D5MS63 A0A2A4JW63 A0A3R9K407 A0A3R9LF12 A0A0L7L495 H1AD93 A0A1L8DZ07 A0A2H1VW29 A0A2W1BC60 A0A194QZM0 A0A385I359 V9VFW4 Q3U139 A0A1E1J9G8 A3KBZ5 H1AD96 P08659 A0A1L8DY75 Q75ZG1 A0A2A4JUE1 A0A194R547 A0A194PUZ5 A3KBZ6 A3KBZ8 A0A3S2NR21 A3KBZ9 A3KBZ7 A0A1Q2T850 A0A1B3TNR9 A0A2H1VGY5 G9M6I9 A0A1Q2U2F7 A0A3F2YLV6
A0A2H1VFT6 A0A2W1C0Z5 A0A2A4JE16 A0A0L7LS68 A0A1B3TNZ3 A0A1B3TP14 A0A2W1BFJ2 A0A2A4JFU1 A0A1B3TNX9 A0A1B3TP12 A0A2A4JCF3 A0A1B3TNZ2 A0A1B3TNX2 A0A1B3TP25 A0A2W1BJK0 A0A1B3TP11 A0A1B3TP22 A0A3S2N8I8 A0A2H1V3V7 A0A1B3TNZ6 A0A2W1BLY3 A0A2A4J5M1 A0A194Q640 A0A1B3TP10 A0A1B3TNZ4 A0A212FBS4 A0A2H1VAR1 A0A2A4JDK9 A0A1B3TP07 A0A2H1VV91 A0A2A4JGT9 A0A2W1BJT9 A0A2H1WV86 A0A1B3TNX3 A0A2H1W204 A0A1B3TNY0 A0A194Q0P0 A0A1B3TNZ0 A0A0N1PKC2 A0A2A4J4G8 A0A1B3TNZ5 A0A194PZW6 A0A2W1BGF2 A0A1B3TNW9 A0A1B3TP01 A0A212FN15 A0A194Q1K5 A0A194R1K6 A0A194R232 A0A1B3TP24 A0A2H1V5T4 A0A2H1V0G3 A0A2H1V5V8 A0A2H1VH10 A0A2H1WMC6 A0A2T6WK65 A0A2A4JKH2 A0A2W1B861 A0A428D0L8 A0A2S3QZT6 D5MS63 A0A2A4JW63 A0A3R9K407 A0A3R9LF12 A0A0L7L495 H1AD93 A0A1L8DZ07 A0A2H1VW29 A0A2W1BC60 A0A194QZM0 A0A385I359 V9VFW4 Q3U139 A0A1E1J9G8 A3KBZ5 H1AD96 P08659 A0A1L8DY75 Q75ZG1 A0A2A4JUE1 A0A194R547 A0A194PUZ5 A3KBZ6 A3KBZ8 A0A3S2NR21 A3KBZ9 A3KBZ7 A0A1Q2T850 A0A1B3TNR9 A0A2H1VGY5 G9M6I9 A0A1Q2U2F7 A0A3F2YLV6
EC Number
1.13.12.7
Pubmed
EMBL
LADJ01029118
KPJ21198.1
KQ459582
KPI98876.1
KQ460878
KPJ11571.1
+ More
KU925757 AOG75382.1 KU925719 AOG75344.1 KU925729 AOG75354.1 ODYU01002332 SOQ39697.1 KZ149898 PZC78636.1 NWSH01001926 PCG69652.1 JTDY01000201 KOB78320.1 KU925742 AOG75367.1 KU925772 AOG75397.1 KZ150081 PZC73879.1 NWSH01001632 PCG70636.1 KU925730 AOG75355.1 KU925758 AOG75383.1 NWSH01001893 PCG69777.1 KU925733 AOG75358.1 KU925720 AOG75345.1 KU925774 AOG75399.1 PZC73875.1 KU925759 AOG75384.1 KU925773 AOG75398.1 RSAL01000205 RVE44356.1 ODYU01000351 SOQ34964.1 KU925747 AOG75372.1 PZC73876.1 NWSH01003054 PCG67036.1 KPI98875.1 KU925762 AOG75387.1 KU925743 AOG75368.1 AGBW02009293 OWR51173.1 ODYU01001291 SOQ37304.1 PCG69778.1 KU925761 AOG75386.1 ODYU01004651 SOQ44755.1 PCG70633.1 KZ150128 PZC73096.1 ODYU01011279 SOQ56917.1 KU925722 AOG75347.1 ODYU01005841 SOQ47125.1 KU925732 AOG75357.1 KPI98873.1 KU925745 AOG75370.1 KPJ21196.1 PCG67037.1 KU925746 AOG75371.1 KPI98872.1 PZC73878.1 KU925721 AOG75346.1 KU925744 AOG75369.1 AGBW02007648 OWR55128.1 KPI98874.1 KPJ11572.1 KPJ11574.1 KU925775 AOG75400.1 ODYU01000839 SOQ36210.1 ODYU01000111 SOQ34327.1 ODYU01000840 SOQ36211.1 ODYU01002515 SOQ40108.1 ODYU01009613 SOQ54167.1 PYJI01000254 PUE89139.1 NWSH01001271 PCG71900.1 KZ150230 PZC72009.1 RJNV01000052 RSI85664.1 PDGH01000119 POB45175.1 AB535101 BAJ07977.1 NWSH01000564 PCG75622.1 RJNQ01000038 RSI74579.1 RJPK01000016 RSJ67583.1 JTDY01003125 KOB70109.1 AB644225 BAL46509.1 GFDF01002418 JAV11666.1 ODYU01004634 SOQ44712.1 PZC72011.1 KQ460890 KPJ10988.1 MH759204 MH759205 MH759206 MH759207 AXY64450.1 KF713491 AHC94771.1 AK155098 AK156297 BAE33661.1 CALQ01001999 CCM20260.1 AB261982 BAF48390.1 MH759021 MH759022 MH759023 MH759024 MH759025 MH759026 MH759027 MH759028 MH759029 MH759030 MH759031 MH759032 MH759033 MH759034 MH759035 MH759036 MH759037 MH759038 MH759039 MH759040 MH759041 MH759042 MH759043 MH759044 MH759045 MH759046 MH759047 MH759048 MH759049 MH759050 MH759051 MH759052 MH759053 MH759054 MH759055 MH759056 MH759057 MH759058 MH759059 MH759060 MH759061 MH759062 MH759063 MH759064 MH759065 MH759066 MH759067 MH759068 MH759069 MH759070 MH759071 MH759072 MH759073 MH759074 MH759075 MH759076 MH759077 MH759078 MH759079 MH759080 MH759081 MH759082 MH759083 MH759084 MH759085 MH759086 MH759087 MH759088 MH759089 MH759090 MH759091 MH759092 MH759093 MH759094 MH759095 MH759096 MH759097 MH759098 MH759099 MH759100 MH759101 MH759102 MH759103 MH759104 MH759105 MH759106 MH759107 MH759108 MH759109 MH759110 MH759111 MH759112 MH759113 MH759114 MH759115 MH759116 MH759117 MH759118 MH759119 MH759120 MH759121 MH759122 MH759123 MH759124 MH759125 MH759126 MH759127 MH759128 MH759129 MH759130 MH759131 MH759132 MH759133 MH759134 MH759135 MH759136 MH759137 MH759138 MH759139 MH759140 MH759141 MH759142 MH759143 MH759144 MH759145 MH759146 MH759147 MH759148 MH759149 MH759150 MH759151 MH759152 MH759153 MH759154 MH759155 MH759156 MH759157 MH759158 MH759159 MH759160 MH759161 MH759162 MH759163 MH759164 MH759165 MH759166 MH759167 MH759168 MH759169 MH759170 MH759171 MH759172 MH759173 MH759174 MH759175 MH759176 MH759177 MH759178 MH759179 MH759180 MH759181 MH759182 MH759183 MH759184 MH759185 MH759186 MH759187 MH759188 MH759189 MH759190 MH759191 MH759192 MH759193 MH759194 MH759195 MH759196 MH759197 MH759198 MH759199 MH759200 MH759201 MH759202 MH759203 MH759208 MH759209 MH759210 AB644228 GEZM01002375 AXY64266.1 BAL46512.1 JAV97291.1 M15077 X84846 X84848 U03687 U89934 U89935 GFDF01002674 JAV11410.1 AB119282 BAD00047.1 PCG75621.1 KPJ10986.1 KQ459593 KPI96564.1 AB261983 BAF48391.1 AB261985 BAF48393.1 RSAL01000342 RVE42388.1 AB261986 BAF48394.1 AB261984 BAF48392.1 LC198321 BAW87787.1 KU670945 AOG75000.1 SOQ40110.1 AB604790 BAL40875.1 LC215694 BAW98153.1
KU925757 AOG75382.1 KU925719 AOG75344.1 KU925729 AOG75354.1 ODYU01002332 SOQ39697.1 KZ149898 PZC78636.1 NWSH01001926 PCG69652.1 JTDY01000201 KOB78320.1 KU925742 AOG75367.1 KU925772 AOG75397.1 KZ150081 PZC73879.1 NWSH01001632 PCG70636.1 KU925730 AOG75355.1 KU925758 AOG75383.1 NWSH01001893 PCG69777.1 KU925733 AOG75358.1 KU925720 AOG75345.1 KU925774 AOG75399.1 PZC73875.1 KU925759 AOG75384.1 KU925773 AOG75398.1 RSAL01000205 RVE44356.1 ODYU01000351 SOQ34964.1 KU925747 AOG75372.1 PZC73876.1 NWSH01003054 PCG67036.1 KPI98875.1 KU925762 AOG75387.1 KU925743 AOG75368.1 AGBW02009293 OWR51173.1 ODYU01001291 SOQ37304.1 PCG69778.1 KU925761 AOG75386.1 ODYU01004651 SOQ44755.1 PCG70633.1 KZ150128 PZC73096.1 ODYU01011279 SOQ56917.1 KU925722 AOG75347.1 ODYU01005841 SOQ47125.1 KU925732 AOG75357.1 KPI98873.1 KU925745 AOG75370.1 KPJ21196.1 PCG67037.1 KU925746 AOG75371.1 KPI98872.1 PZC73878.1 KU925721 AOG75346.1 KU925744 AOG75369.1 AGBW02007648 OWR55128.1 KPI98874.1 KPJ11572.1 KPJ11574.1 KU925775 AOG75400.1 ODYU01000839 SOQ36210.1 ODYU01000111 SOQ34327.1 ODYU01000840 SOQ36211.1 ODYU01002515 SOQ40108.1 ODYU01009613 SOQ54167.1 PYJI01000254 PUE89139.1 NWSH01001271 PCG71900.1 KZ150230 PZC72009.1 RJNV01000052 RSI85664.1 PDGH01000119 POB45175.1 AB535101 BAJ07977.1 NWSH01000564 PCG75622.1 RJNQ01000038 RSI74579.1 RJPK01000016 RSJ67583.1 JTDY01003125 KOB70109.1 AB644225 BAL46509.1 GFDF01002418 JAV11666.1 ODYU01004634 SOQ44712.1 PZC72011.1 KQ460890 KPJ10988.1 MH759204 MH759205 MH759206 MH759207 AXY64450.1 KF713491 AHC94771.1 AK155098 AK156297 BAE33661.1 CALQ01001999 CCM20260.1 AB261982 BAF48390.1 MH759021 MH759022 MH759023 MH759024 MH759025 MH759026 MH759027 MH759028 MH759029 MH759030 MH759031 MH759032 MH759033 MH759034 MH759035 MH759036 MH759037 MH759038 MH759039 MH759040 MH759041 MH759042 MH759043 MH759044 MH759045 MH759046 MH759047 MH759048 MH759049 MH759050 MH759051 MH759052 MH759053 MH759054 MH759055 MH759056 MH759057 MH759058 MH759059 MH759060 MH759061 MH759062 MH759063 MH759064 MH759065 MH759066 MH759067 MH759068 MH759069 MH759070 MH759071 MH759072 MH759073 MH759074 MH759075 MH759076 MH759077 MH759078 MH759079 MH759080 MH759081 MH759082 MH759083 MH759084 MH759085 MH759086 MH759087 MH759088 MH759089 MH759090 MH759091 MH759092 MH759093 MH759094 MH759095 MH759096 MH759097 MH759098 MH759099 MH759100 MH759101 MH759102 MH759103 MH759104 MH759105 MH759106 MH759107 MH759108 MH759109 MH759110 MH759111 MH759112 MH759113 MH759114 MH759115 MH759116 MH759117 MH759118 MH759119 MH759120 MH759121 MH759122 MH759123 MH759124 MH759125 MH759126 MH759127 MH759128 MH759129 MH759130 MH759131 MH759132 MH759133 MH759134 MH759135 MH759136 MH759137 MH759138 MH759139 MH759140 MH759141 MH759142 MH759143 MH759144 MH759145 MH759146 MH759147 MH759148 MH759149 MH759150 MH759151 MH759152 MH759153 MH759154 MH759155 MH759156 MH759157 MH759158 MH759159 MH759160 MH759161 MH759162 MH759163 MH759164 MH759165 MH759166 MH759167 MH759168 MH759169 MH759170 MH759171 MH759172 MH759173 MH759174 MH759175 MH759176 MH759177 MH759178 MH759179 MH759180 MH759181 MH759182 MH759183 MH759184 MH759185 MH759186 MH759187 MH759188 MH759189 MH759190 MH759191 MH759192 MH759193 MH759194 MH759195 MH759196 MH759197 MH759198 MH759199 MH759200 MH759201 MH759202 MH759203 MH759208 MH759209 MH759210 AB644228 GEZM01002375 AXY64266.1 BAL46512.1 JAV97291.1 M15077 X84846 X84848 U03687 U89934 U89935 GFDF01002674 JAV11410.1 AB119282 BAD00047.1 PCG75621.1 KPJ10986.1 KQ459593 KPI96564.1 AB261983 BAF48391.1 AB261985 BAF48393.1 RSAL01000342 RVE42388.1 AB261986 BAF48394.1 AB261984 BAF48392.1 LC198321 BAW87787.1 KU670945 AOG75000.1 SOQ40110.1 AB604790 BAL40875.1 LC215694 BAW98153.1
Proteomes
PRIDE
Interpro
IPR016696
TRAPP-I_su5
+ More
IPR007194 TRAPP_component
IPR000873 AMP-dep_Synth/Lig
IPR025110 AMP-bd_C
IPR024096 NO_sig/Golgi_transp_ligand-bd
IPR020845 AMP-binding_CS
IPR042099 AMP-dep_Synthh-like_sf
IPR027380 Luciferase-like_N_sf
IPR024165 Kan/Strep_kinase
IPR011009 Kinase-like_dom_sf
IPR002575 Aminoglycoside_PTrfase
IPR001254 Trypsin_dom
IPR018114 TRYPSIN_HIS
IPR009003 Peptidase_S1_PA
IPR001314 Peptidase_S1A
IPR033116 TRYPSIN_SER
IPR007194 TRAPP_component
IPR000873 AMP-dep_Synth/Lig
IPR025110 AMP-bd_C
IPR024096 NO_sig/Golgi_transp_ligand-bd
IPR020845 AMP-binding_CS
IPR042099 AMP-dep_Synthh-like_sf
IPR027380 Luciferase-like_N_sf
IPR024165 Kan/Strep_kinase
IPR011009 Kinase-like_dom_sf
IPR002575 Aminoglycoside_PTrfase
IPR001254 Trypsin_dom
IPR018114 TRYPSIN_HIS
IPR009003 Peptidase_S1_PA
IPR001314 Peptidase_S1A
IPR033116 TRYPSIN_SER
Gene 3D
ProteinModelPortal
A0A0N1IQK8
A0A194PZY0
A0A194R6T5
A0A1B3TP04
A0A1B3TNX4
A0A1B3TNY4
+ More
A0A2H1VFT6 A0A2W1C0Z5 A0A2A4JE16 A0A0L7LS68 A0A1B3TNZ3 A0A1B3TP14 A0A2W1BFJ2 A0A2A4JFU1 A0A1B3TNX9 A0A1B3TP12 A0A2A4JCF3 A0A1B3TNZ2 A0A1B3TNX2 A0A1B3TP25 A0A2W1BJK0 A0A1B3TP11 A0A1B3TP22 A0A3S2N8I8 A0A2H1V3V7 A0A1B3TNZ6 A0A2W1BLY3 A0A2A4J5M1 A0A194Q640 A0A1B3TP10 A0A1B3TNZ4 A0A212FBS4 A0A2H1VAR1 A0A2A4JDK9 A0A1B3TP07 A0A2H1VV91 A0A2A4JGT9 A0A2W1BJT9 A0A2H1WV86 A0A1B3TNX3 A0A2H1W204 A0A1B3TNY0 A0A194Q0P0 A0A1B3TNZ0 A0A0N1PKC2 A0A2A4J4G8 A0A1B3TNZ5 A0A194PZW6 A0A2W1BGF2 A0A1B3TNW9 A0A1B3TP01 A0A212FN15 A0A194Q1K5 A0A194R1K6 A0A194R232 A0A1B3TP24 A0A2H1V5T4 A0A2H1V0G3 A0A2H1V5V8 A0A2H1VH10 A0A2H1WMC6 A0A2T6WK65 A0A2A4JKH2 A0A2W1B861 A0A428D0L8 A0A2S3QZT6 D5MS63 A0A2A4JW63 A0A3R9K407 A0A3R9LF12 A0A0L7L495 H1AD93 A0A1L8DZ07 A0A2H1VW29 A0A2W1BC60 A0A194QZM0 A0A385I359 V9VFW4 Q3U139 A0A1E1J9G8 A3KBZ5 H1AD96 P08659 A0A1L8DY75 Q75ZG1 A0A2A4JUE1 A0A194R547 A0A194PUZ5 A3KBZ6 A3KBZ8 A0A3S2NR21 A3KBZ9 A3KBZ7 A0A1Q2T850 A0A1B3TNR9 A0A2H1VGY5 G9M6I9 A0A1Q2U2F7 A0A3F2YLV6
A0A2H1VFT6 A0A2W1C0Z5 A0A2A4JE16 A0A0L7LS68 A0A1B3TNZ3 A0A1B3TP14 A0A2W1BFJ2 A0A2A4JFU1 A0A1B3TNX9 A0A1B3TP12 A0A2A4JCF3 A0A1B3TNZ2 A0A1B3TNX2 A0A1B3TP25 A0A2W1BJK0 A0A1B3TP11 A0A1B3TP22 A0A3S2N8I8 A0A2H1V3V7 A0A1B3TNZ6 A0A2W1BLY3 A0A2A4J5M1 A0A194Q640 A0A1B3TP10 A0A1B3TNZ4 A0A212FBS4 A0A2H1VAR1 A0A2A4JDK9 A0A1B3TP07 A0A2H1VV91 A0A2A4JGT9 A0A2W1BJT9 A0A2H1WV86 A0A1B3TNX3 A0A2H1W204 A0A1B3TNY0 A0A194Q0P0 A0A1B3TNZ0 A0A0N1PKC2 A0A2A4J4G8 A0A1B3TNZ5 A0A194PZW6 A0A2W1BGF2 A0A1B3TNW9 A0A1B3TP01 A0A212FN15 A0A194Q1K5 A0A194R1K6 A0A194R232 A0A1B3TP24 A0A2H1V5T4 A0A2H1V0G3 A0A2H1V5V8 A0A2H1VH10 A0A2H1WMC6 A0A2T6WK65 A0A2A4JKH2 A0A2W1B861 A0A428D0L8 A0A2S3QZT6 D5MS63 A0A2A4JW63 A0A3R9K407 A0A3R9LF12 A0A0L7L495 H1AD93 A0A1L8DZ07 A0A2H1VW29 A0A2W1BC60 A0A194QZM0 A0A385I359 V9VFW4 Q3U139 A0A1E1J9G8 A3KBZ5 H1AD96 P08659 A0A1L8DY75 Q75ZG1 A0A2A4JUE1 A0A194R547 A0A194PUZ5 A3KBZ6 A3KBZ8 A0A3S2NR21 A3KBZ9 A3KBZ7 A0A1Q2T850 A0A1B3TNR9 A0A2H1VGY5 G9M6I9 A0A1Q2U2F7 A0A3F2YLV6
PDB
4G36
E-value=3.39907e-26,
Score=287
Ontologies
GO
PANTHER
Topology
Subcellular location
Peroxisome
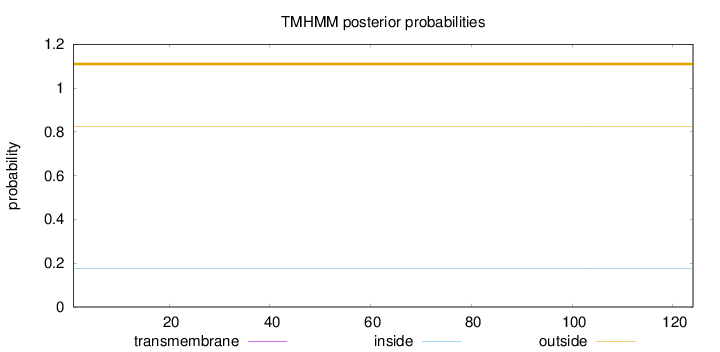
Length:
124
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
0.00027
Exp number, first 60 AAs:
0.00027
Total prob of N-in:
0.17502
outside
1 - 124
Population Genetic Test Statistics
Pi
223.126929
Theta
154.506703
Tajima's D
1.369269
CLR
0.060807
CSRT
0.760161991900405
Interpretation
Uncertain
