Gene
KWMTBOMO08464 Validated by peptides from experiments
Pre Gene Modal
BGIBMGA009393
Annotation
FIB-L_protein_[Bombyx_mori]
Full name
Fibroin light chain
Alternative Name
L-fibroin
PG-1
PG-1
Location in the cell
Extracellular Reliability : 3.752
Sequence
CDS
ATGAAGCCTATATTTTTGGTATTACTCGTCGCTACAAGCGCCTACGCTGCACCATCGGTGACCATCAATCAATACAGTGATAATGAAATTCCACGCGACATTGATGATGGAAAAGCTAGTTCCGTAATCTCACGTGCATGGGACTACGTCGATGACACTGACAAAAGCATCGCCATCCTCAACGTTCAAGAGATCTTGAAGGACATGGCCAGCCAGGGCGATTATGCAAGTCAAGCATCAGCGGTGGCCCAAACCGCCGGAATTATCGCCCATCTATCTGCCGGTATCCCCGGTGATGCCTGTGCAGCCGCTAACGTCATTAACTCTTACACAGACGGCGTCAGGTCCGGAAACTTCGCCGGCTTCAGACAATCTCTCGGTCCCTTCTTCGGACACGTGGGACAAAACTTGAATCTTATCAATCAACTCGTCATCAACCCTGGTCAACTCCGATACTCTGTCGGACCAGCCCTGGGTTGTGCCGGAGGTGGAAGAATCTATGACTTCGAAGCCGCTTGGGATGCAATCTTAGCCAGCAGTGACTCTAGTTTCTTAAATGAAGAGTACTGCATCGTCAAGAGATTGTACAACTCTCGCAACAGCCAAAGCAACAACATCGCTGCCTACATCACCGCTCACTTACTTCCCCCGGTTGCTCAAGTGTTCCACCAATCAGCTGGATCAATCACAGACCTCCTGAGAGGCGTTGGCAACGGTAATGACGCGACCGGTTTAGTTGCTAATGCTCAAAGATATATTGCACAAGCAGCCAGCCAGGTTCACGTCTAA
Protein
MKPIFLVLLVATSAYAAPSVTINQYSDNEIPRDIDDGKASSVISRAWDYVDDTDKSIAILNVQEILKDMASQGDYASQASAVAQTAGIIAHLSAGIPGDACAAANVINSYTDGVRSGNFAGFRQSLGPFFGHVGQNLNLINQLVINPGQLRYSVGPALGCAGGGRIYDFEAAWDAILASSDSSFLNEEYCIVKRLYNSRNSQSNNIAAYITAHLLPPVAQVFHQSAGSITDLLRGVGNGNDATGLVANAQRYIAQAASQVHV
Summary
Description
It is likely that the major role of L-chain is to prevent the retention of H-chain in ER by forming the disulfide linkage.
Subunit
Silk fibroin elementary unit consists in a disulfide-linked heavy and light chain and a p25 glycoprotein in molar ratios of 6:6:1. This results in a complex of approximately 2.3 MDa.
Keywords
Acetylation
Complete proteome
Direct protein sequencing
Disulfide bond
Reference proteome
Secreted
Signal
Silk protein
Feature
chain Fibroin light chain
Uniprot
Pubmed
EMBL
X17291
CK545649
CK545775
M76430
HQ116534
ADM35104.1
+ More
AY078388 AAL83649.1 AB001820 BAB39499.1 AF541967 AAN63945.1 AY026058 AAK13024.1 AB001824 BAB39503.1 KQ460144 KPJ17452.1 JX456150 AFS32690.1 AB001822 BAB39501.1 S77817 NWSH01000381 PCG76817.1 KF669645 AHB81536.1 GQ901975 ACX50392.1 KQ459337 KPJ01426.1 RSAL01000059 RVE49691.1 AGBW02013327 OWR43595.1 AB195977 BAE97693.1 GU180667 ADI52841.1 GU180670 ADI52844.1 GU180671 ADI52845.1 GU180668 ADI52842.1 GU180672 ADI52846.1 GU180666 ADI52840.1 GU180674 ADI52848.1 GU180673 ADI52847.1 GU180669 ADI52843.1 GU180675 ADI52849.1
AY078388 AAL83649.1 AB001820 BAB39499.1 AF541967 AAN63945.1 AY026058 AAK13024.1 AB001824 BAB39503.1 KQ460144 KPJ17452.1 JX456150 AFS32690.1 AB001822 BAB39501.1 S77817 NWSH01000381 PCG76817.1 KF669645 AHB81536.1 GQ901975 ACX50392.1 KQ459337 KPJ01426.1 RSAL01000059 RVE49691.1 AGBW02013327 OWR43595.1 AB195977 BAE97693.1 GU180667 ADI52841.1 GU180670 ADI52844.1 GU180671 ADI52845.1 GU180668 ADI52842.1 GU180672 ADI52846.1 GU180666 ADI52840.1 GU180674 ADI52848.1 GU180673 ADI52847.1 GU180669 ADI52843.1 GU180675 ADI52849.1
Proteomes
Pfam
PF05849 L-fibroin
Interpro
IPR008660
L-fibroin
ProteinModelPortal
Ontologies
GO
Topology
Subcellular location
Secreted
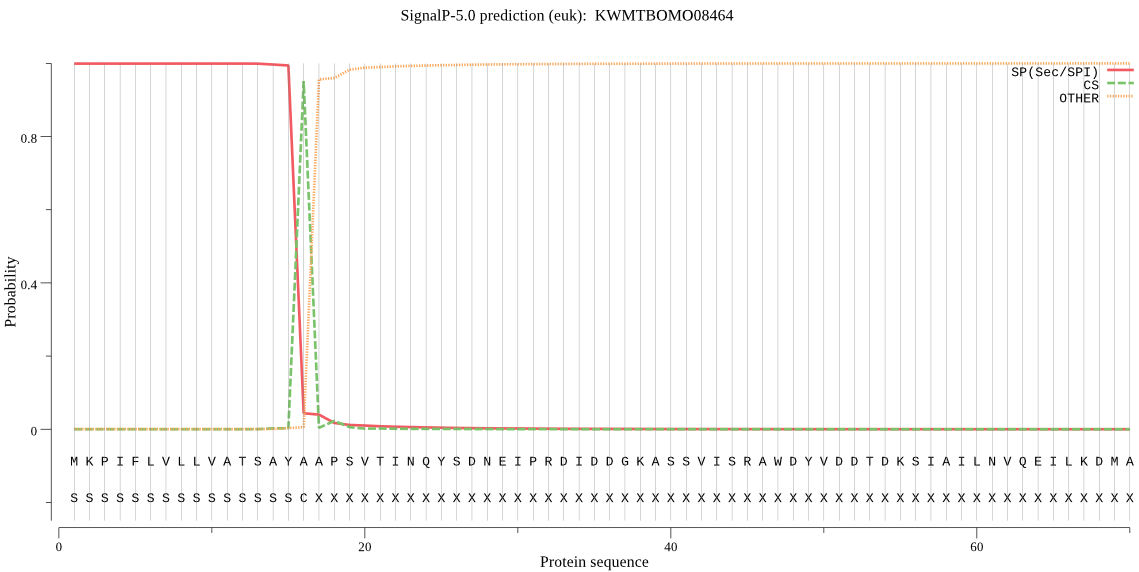
SignalP
Position: 1 - 16,
Likelihood: 0.999024
Length:
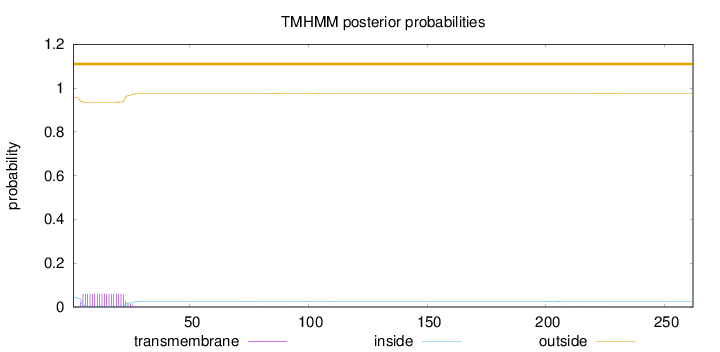
262
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
1.24129
Exp number, first 60 AAs:
1.18211
Total prob of N-in:
0.04342
outside
1 - 262
Population Genetic Test Statistics
Pi
239.014506
Theta
159.823903
Tajima's D
1.658423
CLR
0.205713
CSRT
0.820058997050148
Interpretation
Uncertain
