Gene
KWMTBOMO06256
Pre Gene Modal
BGIBMGA001628
Annotation
PREDICTED:_zinc_finger_protein_484-like_isoform_X2_[Bombyx_mori]
Full name
Zinc finger protein
Transcription factor
Location in the cell
Nuclear Reliability : 4.437
Sequence
CDS
ATGTCGCAAGATGGTCCCATTGTTTTCGTGGATGTAGCTACTGAATACAAAAATGTATATACACCCGAAGAACCTGTTACGGAAACAGTAGCCGTCCAGAACAACACCAATACGAGTAGCATCCAGAATTTCAAGTGTAATGAGTGCAATAGAACCGTAGACAGTGAAGAGAAATTAAGTCAACACATACGGAGGGCACACCGTGGAGAAAATGCTTTTCAATGCTCGATGTGTCAATACTCGACATACAATAAAAGTGGATTCGAAGAACATGTTAGGATACATCAAGGTATAAAACCATTTAAATGCTCCTACTGCCCTTATAGGAGCGTTTCGAAGAAATACGCCAAGAAGCACGAACTGATCCACCGGCCCGATAACCCGTTGAGATGTCAGCACTGTCCTTACATAGCGCGGAACTATCGCATGTTGAATTCCCACCAGAAGAAGTTGCACAGCAACGAGAAACAACGGGTTATCATCGCGTGCACGTTGTGCGGGAAGAAATTCGACGATATGAACGTTTATTTCGCTCACAAGAAACGCAGGAAGCAGTGTGATGAGTGCGGTCTAGAGGTTTGTCATCAGGCGATGAACAGGCACAAACACGAGAAACACGGCGTGAAAAGAAAAGTTCGCGGCAAAGACGTTTTCACATGTAAAATATGCGAATGGTCTTCGGTAAATAAAGTTAAGGTTTTGTTGCACATCATTCATCACCCCGAACAGGATTTGAGCGATTGCAGTTTCGATTTGAGCGTTCTGAAGAAATGTCAAATTTTGCCGCCATAA
Protein
MSQDGPIVFVDVATEYKNVYTPEEPVTETVAVQNNTNTSSIQNFKCNECNRTVDSEEKLSQHIRRAHRGENAFQCSMCQYSTYNKSGFEEHVRIHQGIKPFKCSYCPYRSVSKKYAKKHELIHRPDNPLRCQHCPYIARNYRMLNSHQKKLHSNEKQRVIIACTLCGKKFDDMNVYFAHKKRRKQCDECGLEVCHQAMNRHKHEKHGVKRKVRGKDVFTCKICEWSSVNKVKVLLHIIHHPEQDLSDCSFDLSVLKKCQILPP
Summary
Uniprot
A0A2W1BKI9
A0A2A4JNM4
A0A2H1V402
A0A212F495
A0A194Q5W1
I4DPB5
+ More
A0A1B6CRV2 T1HYQ4 C3XWY4 C3XWX7 A0A023F763 C3XWZ5 C3YHN1 A0A146QN68 C3YTR5 H0WF80 A0A3B5L068 A0A3Q2PT12 C3ZSM4 A0A146QMY2 C3Z011 A0A146PI22 A0A3Q2ZDK0 A0A146STC1 A0A224XSR3 C3YHK2 C3XWY0 C3YM59 H2M5V8 C3YHP7 C3YMG2 Q07G26 C3YQQ7 C3YHR1 G1LK80 T1ICU5 C3YHM9 A0A087XJR5 C3ZP01 C3YHJ9 A0A146Q5F9 C3YHK4 C3YHK6 C3ZRA1 A0A087XDY2 C3YFF8 A0A1B6CP67 A0A0L8FHE3 X1WDJ1 C3YQG4 C3XSP6 C3YHJ2 A0A0A9YQJ8 A0A1A8U672 C3YHM6 C3ZY74 C4A0P9 C3ZYL2 C3YHS4 A0A1A8Q847 C3Z094 C3YHK1 C3ZR96 A0A224XLP4 A0A087X5E0 F7FWV4 C3YHY0 A0A0A9WU59 C3YHN4 C3YUM4 C3XWY3 C3ZY39 C3XXA9 C3YZZ2 C3YHM7 C3YHL0 A0A087XPQ2 C3YHK8 C3YHL8 C3YQQ4 C4A0P7 C3ZL42 I3ISW0 C3YNE1 C3YHP4 A0A0K8SYG2 A0A2R9YJN5 A0A2G9RXT3 I3KI16 M7AI09 A0A069DTB1 A0A1S3WGP8
A0A1B6CRV2 T1HYQ4 C3XWY4 C3XWX7 A0A023F763 C3XWZ5 C3YHN1 A0A146QN68 C3YTR5 H0WF80 A0A3B5L068 A0A3Q2PT12 C3ZSM4 A0A146QMY2 C3Z011 A0A146PI22 A0A3Q2ZDK0 A0A146STC1 A0A224XSR3 C3YHK2 C3XWY0 C3YM59 H2M5V8 C3YHP7 C3YMG2 Q07G26 C3YQQ7 C3YHR1 G1LK80 T1ICU5 C3YHM9 A0A087XJR5 C3ZP01 C3YHJ9 A0A146Q5F9 C3YHK4 C3YHK6 C3ZRA1 A0A087XDY2 C3YFF8 A0A1B6CP67 A0A0L8FHE3 X1WDJ1 C3YQG4 C3XSP6 C3YHJ2 A0A0A9YQJ8 A0A1A8U672 C3YHM6 C3ZY74 C4A0P9 C3ZYL2 C3YHS4 A0A1A8Q847 C3Z094 C3YHK1 C3ZR96 A0A224XLP4 A0A087X5E0 F7FWV4 C3YHY0 A0A0A9WU59 C3YHN4 C3YUM4 C3XWY3 C3ZY39 C3XXA9 C3YZZ2 C3YHM7 C3YHL0 A0A087XPQ2 C3YHK8 C3YHL8 C3YQQ4 C4A0P7 C3ZL42 I3ISW0 C3YNE1 C3YHP4 A0A0K8SYG2 A0A2R9YJN5 A0A2G9RXT3 I3KI16 M7AI09 A0A069DTB1 A0A1S3WGP8
EC Number
2.1.1.-
Pubmed
EMBL
KZ150084
PZC73797.1
NWSH01000911
PCG73571.1
ODYU01000561
SOQ35529.1
+ More
AGBW02010410 OWR48568.1 KQ459463 KPJ00395.1 AK403510 BAM19755.1 GEDC01021201 JAS16097.1 ACPB03021784 GG666471 EEN67244.1 EEN67237.1 GBBI01001678 JAC17034.1 EEN67255.1 GG666514 EEN60018.1 GCES01129116 JAQ57206.1 GG666552 EEN56192.1 BX571813 BX908799 GG666674 EEN44426.1 GCES01129115 JAQ57207.1 GG666567 EEN54070.1 GCES01142804 JAQ43518.1 GCES01102471 JAQ83851.1 GFTR01005263 JAW11163.1 EEN59989.1 EEN67240.1 GG666529 EEN58653.1 EEN60034.1 EEN58756.1 CR848360 CAL49420.1 GG666543 EEN57413.1 EEN60048.1 ACTA01172268 ACPB03016510 EEN60016.1 AYCK01022746 AYCK01022747 GG666654 EEN45736.1 EEN59986.1 GCES01135432 JAQ50890.1 EEN59991.1 EEN59993.1 GG666664 EEN44974.1 AYCK01018735 AYCK01018736 AYCK01018737 GG666509 EEN60921.1 GEDC01022090 GEDC01016829 GEDC01015516 JAS15208.1 JAS20469.1 JAS21782.1 KQ432245 KOF62831.1 CU928013 GG666542 EEN57494.1 GG666459 EEN68967.1 EEN59979.1 GBHO01008282 JAG35322.1 HADY01021620 HAEJ01002624 SBS43081.1 EEN60013.1 GG666722 EEN42505.1 GG666818 EEN41634.1 GG666732 EEN42359.1 EEN60061.1 HAEH01010342 SBR89457.1 GG666568 EEN53942.1 EEN59988.1 EEN44969.1 GFTR01004422 JAW12004.1 AYCK01020751 AYCK01020752 AYCK01020753 AYCK01020754 EEN60117.1 GBHO01043930 GBHO01043929 GBHO01035214 JAF99673.1 JAF99674.1 JAG08390.1 EEN60021.1 GG666554 EEN55925.1 EEN67243.1 GG666721 EEN42521.1 EEN67369.1 EEN54051.1 EEN60014.1 EEN59997.1 AYCK01011382 EEN59995.1 EEN60005.1 EEN57410.1 EEN41632.1 GG666640 EEN46724.1 FP102097 GG666533 EEN58247.1 EEN60031.1 GBRD01007499 GBRD01007498 JAG58323.1 BX649516 KV933395 PIO32021.1 AERX01044840 AERX01044841 AERX01044842 AERX01044843 AERX01044844 KB604988 EMP23954.1 GBGD01002007 JAC86882.1
AGBW02010410 OWR48568.1 KQ459463 KPJ00395.1 AK403510 BAM19755.1 GEDC01021201 JAS16097.1 ACPB03021784 GG666471 EEN67244.1 EEN67237.1 GBBI01001678 JAC17034.1 EEN67255.1 GG666514 EEN60018.1 GCES01129116 JAQ57206.1 GG666552 EEN56192.1 BX571813 BX908799 GG666674 EEN44426.1 GCES01129115 JAQ57207.1 GG666567 EEN54070.1 GCES01142804 JAQ43518.1 GCES01102471 JAQ83851.1 GFTR01005263 JAW11163.1 EEN59989.1 EEN67240.1 GG666529 EEN58653.1 EEN60034.1 EEN58756.1 CR848360 CAL49420.1 GG666543 EEN57413.1 EEN60048.1 ACTA01172268 ACPB03016510 EEN60016.1 AYCK01022746 AYCK01022747 GG666654 EEN45736.1 EEN59986.1 GCES01135432 JAQ50890.1 EEN59991.1 EEN59993.1 GG666664 EEN44974.1 AYCK01018735 AYCK01018736 AYCK01018737 GG666509 EEN60921.1 GEDC01022090 GEDC01016829 GEDC01015516 JAS15208.1 JAS20469.1 JAS21782.1 KQ432245 KOF62831.1 CU928013 GG666542 EEN57494.1 GG666459 EEN68967.1 EEN59979.1 GBHO01008282 JAG35322.1 HADY01021620 HAEJ01002624 SBS43081.1 EEN60013.1 GG666722 EEN42505.1 GG666818 EEN41634.1 GG666732 EEN42359.1 EEN60061.1 HAEH01010342 SBR89457.1 GG666568 EEN53942.1 EEN59988.1 EEN44969.1 GFTR01004422 JAW12004.1 AYCK01020751 AYCK01020752 AYCK01020753 AYCK01020754 EEN60117.1 GBHO01043930 GBHO01043929 GBHO01035214 JAF99673.1 JAF99674.1 JAG08390.1 EEN60021.1 GG666554 EEN55925.1 EEN67243.1 GG666721 EEN42521.1 EEN67369.1 EEN54051.1 EEN60014.1 EEN59997.1 AYCK01011382 EEN59995.1 EEN60005.1 EEN57410.1 EEN41632.1 GG666640 EEN46724.1 FP102097 GG666533 EEN58247.1 EEN60031.1 GBRD01007499 GBRD01007498 JAG58323.1 BX649516 KV933395 PIO32021.1 AERX01044840 AERX01044841 AERX01044842 AERX01044843 AERX01044844 KB604988 EMP23954.1 GBGD01002007 JAC86882.1
Proteomes
Interpro
ProteinModelPortal
A0A2W1BKI9
A0A2A4JNM4
A0A2H1V402
A0A212F495
A0A194Q5W1
I4DPB5
+ More
A0A1B6CRV2 T1HYQ4 C3XWY4 C3XWX7 A0A023F763 C3XWZ5 C3YHN1 A0A146QN68 C3YTR5 H0WF80 A0A3B5L068 A0A3Q2PT12 C3ZSM4 A0A146QMY2 C3Z011 A0A146PI22 A0A3Q2ZDK0 A0A146STC1 A0A224XSR3 C3YHK2 C3XWY0 C3YM59 H2M5V8 C3YHP7 C3YMG2 Q07G26 C3YQQ7 C3YHR1 G1LK80 T1ICU5 C3YHM9 A0A087XJR5 C3ZP01 C3YHJ9 A0A146Q5F9 C3YHK4 C3YHK6 C3ZRA1 A0A087XDY2 C3YFF8 A0A1B6CP67 A0A0L8FHE3 X1WDJ1 C3YQG4 C3XSP6 C3YHJ2 A0A0A9YQJ8 A0A1A8U672 C3YHM6 C3ZY74 C4A0P9 C3ZYL2 C3YHS4 A0A1A8Q847 C3Z094 C3YHK1 C3ZR96 A0A224XLP4 A0A087X5E0 F7FWV4 C3YHY0 A0A0A9WU59 C3YHN4 C3YUM4 C3XWY3 C3ZY39 C3XXA9 C3YZZ2 C3YHM7 C3YHL0 A0A087XPQ2 C3YHK8 C3YHL8 C3YQQ4 C4A0P7 C3ZL42 I3ISW0 C3YNE1 C3YHP4 A0A0K8SYG2 A0A2R9YJN5 A0A2G9RXT3 I3KI16 M7AI09 A0A069DTB1 A0A1S3WGP8
A0A1B6CRV2 T1HYQ4 C3XWY4 C3XWX7 A0A023F763 C3XWZ5 C3YHN1 A0A146QN68 C3YTR5 H0WF80 A0A3B5L068 A0A3Q2PT12 C3ZSM4 A0A146QMY2 C3Z011 A0A146PI22 A0A3Q2ZDK0 A0A146STC1 A0A224XSR3 C3YHK2 C3XWY0 C3YM59 H2M5V8 C3YHP7 C3YMG2 Q07G26 C3YQQ7 C3YHR1 G1LK80 T1ICU5 C3YHM9 A0A087XJR5 C3ZP01 C3YHJ9 A0A146Q5F9 C3YHK4 C3YHK6 C3ZRA1 A0A087XDY2 C3YFF8 A0A1B6CP67 A0A0L8FHE3 X1WDJ1 C3YQG4 C3XSP6 C3YHJ2 A0A0A9YQJ8 A0A1A8U672 C3YHM6 C3ZY74 C4A0P9 C3ZYL2 C3YHS4 A0A1A8Q847 C3Z094 C3YHK1 C3ZR96 A0A224XLP4 A0A087X5E0 F7FWV4 C3YHY0 A0A0A9WU59 C3YHN4 C3YUM4 C3XWY3 C3ZY39 C3XXA9 C3YZZ2 C3YHM7 C3YHL0 A0A087XPQ2 C3YHK8 C3YHL8 C3YQQ4 C4A0P7 C3ZL42 I3ISW0 C3YNE1 C3YHP4 A0A0K8SYG2 A0A2R9YJN5 A0A2G9RXT3 I3KI16 M7AI09 A0A069DTB1 A0A1S3WGP8
PDB
5UND
E-value=2.53999e-11,
Score=163
Ontologies
GO
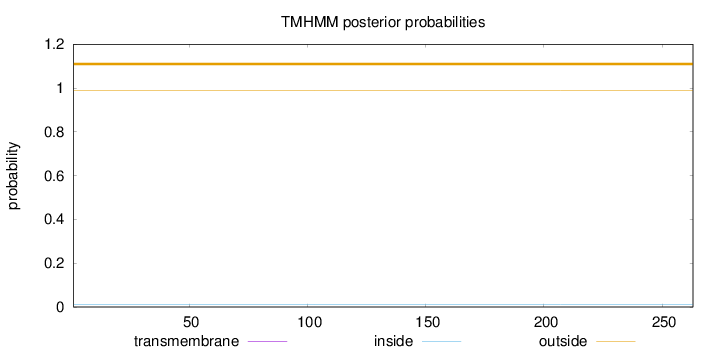
Topology
Length:
263
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
0.0002
Exp number, first 60 AAs:
0
Total prob of N-in:
0.01154
outside
1 - 263
Population Genetic Test Statistics
Pi
161.823832
Theta
152.417427
Tajima's D
0.074651
CLR
2.052918
CSRT
0.391080445977701
Interpretation
Uncertain
