Gene
KWMTBOMO05820 Validated by peptides from experiments
Pre Gene Modal
BGIBMGA006907
Annotation
isocitrate_dehydrogenase_[Bombyx_mori]
Full name
Isocitrate dehydrogenase [NADP]
Location in the cell
Cytoplasmic Reliability : 4.682
Sequence
CDS
ATGTCTAAAATAAAGGCTGGACCTGTTGTTGACATCCTTGGTGATGAAATGACCAGAATAATCTGGGATCTTATCAAAGAAAAGCTGATTCTTCCTTTCTTGGACATTGAATTACATGTCTACGACTTGGGTATGGAAAATCGTGATAAGACTGATGATCAAGTTACAATTGATTGTGCTGAGGCCATAAAGAAATACAATGTGGGCATCAAATGTGCGACCATAACTCCTGATGAGAAGAGAGTTGAAGAATTTAAACTGAAGAAAATGTGGAAAAGCCCTAATGGGACCATCCGTAATATTCTTGGTGGTACTGTTTTCAGGGAGGCTATTATTTGCAAGAATATACCGAGACTTGTAACAGGGTGGGACAAACCCATCATCATTGGACGTCATGCTCATGCTGATCAATACAAAGCAACTGACTTTGTTGTCCCAGGTGCTGGTACTCTTGAAATAATCTTCAAACCTGAATCTGGTGAGGCAATAAAACATGTAGTTCATGAGTACAAGGGTGCAGGTGTAGCACTGGCTATGTTTAACACTGATGCATCTATTATTGACTTTGCTCATTCTTCATTCAAGTTTGCTTTGGACAGAAAATACCCTCTGTACTTAAGCACTAAGAATACAATTCTCAAGAAATACGATGGTCGTTTCAAAGACATCTTTCAAGATATTTATGACAGGGAGTACAAGAAACAGTTTGAGGATGCTGGTATCTGGTATGAACACAGGCTAATTGATGATATGGTGGCTTATGCTATGAAATCAGAGGGTGGATTTGTCTGGGCCTGCAAAAATTATGATGGAGACGTACAATCTGATAGTGTTGCTCAGGGCTATGGGTCACTGGGCCTCATGACATCTGTTCTCATTTGTCCTGATGGCAAAACAGTTGAGGCTGAAGCTGCCCATGGCACTGTTACACGTCATTTCAGATTCTATCAGCAAGGAAAAGAGACATCCACCAATCCAATTGCTTCAATTTTTGCTTGGACTAGAGGTTTGTTGCATCGTGCTAAATTAGACAACAATGATGCCTTAAAGAACTTTGCTGAGACTCTGGAGAAGGTCTGCATTGAAACTATTGAATCTGGAATTATGACTAAAGACCTTGCCATTTGCATCAAAGGTATGAACAATGTCAAAAGATCAGACTACTATGAAACATTCGAATTCATGGATAAACTAGCCGAAAACCTGAAGGTTGCTCTAGGACAATGA
Protein
MSKIKAGPVVDILGDEMTRIIWDLIKEKLILPFLDIELHVYDLGMENRDKTDDQVTIDCAEAIKKYNVGIKCATITPDEKRVEEFKLKKMWKSPNGTIRNILGGTVFREAIICKNIPRLVTGWDKPIIIGRHAHADQYKATDFVVPGAGTLEIIFKPESGEAIKHVVHEYKGAGVALAMFNTDASIIDFAHSSFKFALDRKYPLYLSTKNTILKKYDGRFKDIFQDIYDREYKKQFEDAGIWYEHRLIDDMVAYAMKSEGGFVWACKNYDGDVQSDSVAQGYGSLGLMTSVLICPDGKTVEAEAAHGTVTRHFRFYQQGKETSTNPIASIFAWTRGLLHRAKLDNNDALKNFAETLEKVCIETIESGIMTKDLAICIKGMNNVKRSDYYETFEFMDKLAENLKVALGQ
Summary
Catalytic Activity
isocitrate + NADP(+) = 2-oxoglutarate + CO2 + NADPH
Similarity
Belongs to the isocitrate and isopropylmalate dehydrogenases family.
Uniprot
Q2F681
H9JBL2
A0A2H1WVV4
A0A2A4JR90
A0A3S2NRY6
A0A0L7LLS3
+ More
A0A212F0Y6 S4NRX2 I4DJJ5 A0A194QWA6 A0A1B0CTD9 A0A1L8E0K3 A0A1L8E0D2 D6X0W4 A0A0K8TU47 A0A1I8N1J5 A0A1L8EDV4 T1P7N4 A0A1I8P3Z8 A0A0L0CQS9 A0A1L8E9W3 A0A1I8P3X6 A0A1B6GLY1 A0A1Y1MCE9 A0A1B0D371 A0A034VY52 A0A0K8V5S9 A0A034VXI0 A0A0C9RE92 A0A0K8VIU0 A0A0K8UQX7 A0A1W4XI19 A0A0K8UYM3 A0A1B6C244 A0A0A1XSM3 V5GLZ3 A0A0C5CP30 A0A0C5CKF5 A0A2P8ZLT9 A0A0C5CP10 A0A0C5CKC5 A0A0C5CGB8 A0A0C5D1E6 W8BYY5 A0A0C5CNT3 A0A1W5YLL3 A0A0C5CP36 Q1HQW5 A0A0C5CKD6 A0A0C5CKU5 A0A0C5CKG7 A0A0C5CNZ5 A0A0C5D1K8 A0A2S2NE75 A0A0C5D1J6 A0A0C5D1L2 A0A0C5CP33 A0A0C5CP39 A0A0C5CKV9 A0A182H172 A0A0C5CKG6 A0A2J7PE17 A0A0C5CGG5 A0A1Q3FC67 A0A2S2R9R6 A0A2H8TZ88 A0A2R7WYM0 Q09JZ8 A0A0C5QRX3 B0VZM6 A0A1S3DMV7 A0A0C5CP27 A0A2Z5RE48 A0A1A9VYM5 A0A1B0AYA6 B4QLN4 A0A0J9RST1 A0A0J9RRM2 D3TLG3 A0A1A9XWJ2 A0A0A9WI82 A0A1A9WRA5 A0A0J9RR79 A0A0P8XU25 B3M5J6 B4N6D3 B7Z0E0 A0A067QHM4 A0A182PMH2 Q7KUB1 A0A0P9AJT6 E2BBR3 A0A0R1DVD9 Q9VSI6 A0A0J9RRC2 B4PG37 B4HJH5 A0A0R1DV11 V5SKH7 A0A0Q5U2W9
A0A212F0Y6 S4NRX2 I4DJJ5 A0A194QWA6 A0A1B0CTD9 A0A1L8E0K3 A0A1L8E0D2 D6X0W4 A0A0K8TU47 A0A1I8N1J5 A0A1L8EDV4 T1P7N4 A0A1I8P3Z8 A0A0L0CQS9 A0A1L8E9W3 A0A1I8P3X6 A0A1B6GLY1 A0A1Y1MCE9 A0A1B0D371 A0A034VY52 A0A0K8V5S9 A0A034VXI0 A0A0C9RE92 A0A0K8VIU0 A0A0K8UQX7 A0A1W4XI19 A0A0K8UYM3 A0A1B6C244 A0A0A1XSM3 V5GLZ3 A0A0C5CP30 A0A0C5CKF5 A0A2P8ZLT9 A0A0C5CP10 A0A0C5CKC5 A0A0C5CGB8 A0A0C5D1E6 W8BYY5 A0A0C5CNT3 A0A1W5YLL3 A0A0C5CP36 Q1HQW5 A0A0C5CKD6 A0A0C5CKU5 A0A0C5CKG7 A0A0C5CNZ5 A0A0C5D1K8 A0A2S2NE75 A0A0C5D1J6 A0A0C5D1L2 A0A0C5CP33 A0A0C5CP39 A0A0C5CKV9 A0A182H172 A0A0C5CKG6 A0A2J7PE17 A0A0C5CGG5 A0A1Q3FC67 A0A2S2R9R6 A0A2H8TZ88 A0A2R7WYM0 Q09JZ8 A0A0C5QRX3 B0VZM6 A0A1S3DMV7 A0A0C5CP27 A0A2Z5RE48 A0A1A9VYM5 A0A1B0AYA6 B4QLN4 A0A0J9RST1 A0A0J9RRM2 D3TLG3 A0A1A9XWJ2 A0A0A9WI82 A0A1A9WRA5 A0A0J9RR79 A0A0P8XU25 B3M5J6 B4N6D3 B7Z0E0 A0A067QHM4 A0A182PMH2 Q7KUB1 A0A0P9AJT6 E2BBR3 A0A0R1DVD9 Q9VSI6 A0A0J9RRC2 B4PG37 B4HJH5 A0A0R1DV11 V5SKH7 A0A0Q5U2W9
EC Number
1.1.1.42
Pubmed
19121390
26227816
22118469
23622113
22651552
26354079
+ More
18362917 19820115 26369729 25315136 26108605 28004739 25348373 25830018 25470210 29403074 24495485 17204158 17510324 26483478 25702953 26760975 17994087 22936249 20353571 25401762 26823975 18057021 10731132 12537568 12537572 12537573 12537574 16110336 17569856 17569867 24845553 20798317 17550304 26109357 26109356
18362917 19820115 26369729 25315136 26108605 28004739 25348373 25830018 25470210 29403074 24495485 17204158 17510324 26483478 25702953 26760975 17994087 22936249 20353571 25401762 26823975 18057021 10731132 12537568 12537572 12537573 12537574 16110336 17569856 17569867 24845553 20798317 17550304 26109357 26109356
EMBL
DQ311191
KM027251
KM027252
ABD36136.1
AIU94620.1
AIU94621.1
+ More
BABH01024482 ODYU01011457 SOQ57199.1 NWSH01000730 PCG74547.1 RSAL01000003 RVE54841.1 JTDY01000658 KOB76299.1 AGBW02011011 OWR47392.1 GAIX01014292 JAA78268.1 AK401463 KQ459302 BAM18085.1 KPJ01762.1 KQ461175 KPJ07851.1 AJWK01027438 GFDF01001824 JAV12260.1 GFDF01001896 JAV12188.1 KQ971372 EFA09539.1 GDAI01000128 JAI17475.1 GFDG01001899 JAV16900.1 KA644621 AFP59250.1 JRES01000036 KNC34713.1 GFDG01003229 JAV15570.1 GECZ01006426 JAS63343.1 GEZM01037003 GEZM01037002 JAV82270.1 AJVK01023348 AJVK01023349 AJVK01023350 AJVK01023351 GAKP01012162 GAKP01012161 JAC46790.1 GDHF01018042 JAI34272.1 GAKP01012160 JAC46792.1 GBYB01011477 JAG81244.1 GDHF01027592 GDHF01016721 GDHF01013834 JAI24722.1 JAI35593.1 JAI38480.1 GDHF01023594 JAI28720.1 GDHF01020874 JAI31440.1 GEDC01029712 JAS07586.1 GBXI01014999 GBXI01000271 JAC99292.1 JAD14021.1 GALX01003372 JAB65094.1 KJ371291 AJO53481.1 KJ371290 AJO53480.1 PYGN01000020 PSN57464.1 KJ371266 AJO53456.1 KJ371166 KJ371167 KJ371168 KJ371169 KJ371170 KJ371171 KJ371172 KJ371173 KJ371174 KJ371175 KJ371176 KJ371177 KJ371178 KJ371179 KJ371180 KJ371181 KJ371182 KJ371183 KJ371184 KJ371185 KJ371186 KJ371187 KJ371188 KJ371190 KJ371191 KJ371192 KJ371194 KJ371195 KJ371196 KJ371198 KJ371199 KJ371200 KJ371201 KJ371202 KJ371203 KJ371204 KJ371205 KJ371207 KJ371208 KJ371209 KJ371210 KJ371211 KJ371212 KJ371213 KJ371214 KJ371215 KJ371216 KJ371217 KJ371218 KJ371219 KJ371220 KJ371221 KJ371222 KJ371224 KJ371225 KJ371226 KJ371227 KJ371228 KJ371229 KJ371230 KJ371231 KJ371232 KJ371234 KJ371235 KJ371237 KJ371240 KJ371242 KJ371243 KJ371244 KJ371245 KJ371246 KJ371247 KJ371248 KJ371249 KJ371250 KJ371251 KJ371252 KJ371253 KJ371254 KJ371255 KJ371256 KJ371257 KJ371258 KJ371259 KJ371260 KJ371261 KJ371262 KJ371263 KJ371264 KJ371265 KJ371267 KJ371268 KJ371269 KJ371271 KJ371272 KJ371273 KJ371274 KJ371275 KJ371276 KJ371277 KJ371278 KJ371279 KJ371280 KJ371281 KJ371282 KJ371283 KJ371284 KJ371285 AJO53356.1 AJO53357.1 AJO53358.1 AJO53359.1 AJO53360.1 AJO53361.1 AJO53362.1 AJO53363.1 AJO53364.1 AJO53365.1 AJO53366.1 AJO53367.1 AJO53368.1 AJO53369.1 AJO53370.1 AJO53371.1 AJO53372.1 AJO53373.1 AJO53374.1 AJO53375.1 AJO53376.1 AJO53377.1 AJO53378.1 AJO53380.1 AJO53381.1 AJO53382.1 AJO53384.1 AJO53385.1 AJO53386.1 AJO53388.1 AJO53389.1 AJO53390.1 AJO53391.1 AJO53392.1 AJO53393.1 AJO53394.1 AJO53395.1 AJO53397.1 AJO53398.1 AJO53399.1 AJO53400.1 AJO53401.1 AJO53402.1 AJO53403.1 AJO53404.1 AJO53405.1 AJO53406.1 AJO53407.1 AJO53408.1 AJO53409.1 AJO53410.1 AJO53411.1 AJO53412.1 AJO53414.1 AJO53415.1 AJO53416.1 AJO53417.1 AJO53418.1 AJO53419.1 AJO53420.1 AJO53421.1 AJO53422.1 AJO53424.1 AJO53425.1 AJO53427.1 AJO53430.1 AJO53432.1 AJO53433.1 AJO53434.1 AJO53435.1 AJO53436.1 AJO53437.1 AJO53438.1 AJO53439.1 AJO53440.1 AJO53441.1 AJO53442.1 AJO53443.1 AJO53444.1 AJO53445.1 AJO53446.1 AJO53447.1 AJO53448.1 AJO53449.1 AJO53450.1 AJO53451.1 AJO53452.1 AJO53453.1 AJO53454.1 AJO53455.1 AJO53457.1 AJO53458.1 AJO53459.1 AJO53461.1 AJO53462.1 AJO53463.1 AJO53464.1 AJO53465.1 AJO53466.1 AJO53467.1 AJO53468.1 AJO53469.1 AJO53470.1 AJO53471.1 AJO53472.1 AJO53473.1 AJO53474.1 AJO53475.1 KJ371238 KJ371239 AJO53428.1 AJO53429.1 KJ371223 AJO53413.1 GAMC01004653 GAMC01004652 JAC01904.1 KJ371206 KJ371233 KJ371236 AJO53396.1 AJO53423.1 AJO53426.1 KX147675 ARI45079.1 KJ371300 KJ371301 AJO53490.1 AJO53491.1 DQ440329 CH477200 ABF18362.1 EAT48221.1 KJ371270 AJO53460.1 KJ371297 KJ371299 AJO53487.1 AJO53489.1 KJ371189 KJ371193 KJ371197 AJO53379.1 AJO53383.1 AJO53387.1 KJ371241 AJO53431.1 KJ371308 KJ371309 KJ371310 KJ371311 AJO53498.1 AJO53499.1 AJO53500.1 AJO53501.1 GGMR01002457 MBY15076.1 KJ371292 KJ371293 KJ371294 KJ371295 AJO53482.1 AJO53483.1 AJO53484.1 AJO53485.1 KJ371313 KJ371315 AJO53503.1 AJO53505.1 KJ371296 KJ371298 AJO53486.1 AJO53488.1 KJ371302 KJ371303 KJ371304 KJ371306 AJO53492.1 AJO53493.1 AJO53494.1 AJO53496.1 KJ371312 KJ371314 AJO53502.1 AJO53504.1 JXUM01023082 JXUM01023083 JXUM01023084 JXUM01023085 JXUM01023086 JXUM01023087 KJ371305 KJ371307 AJO53495.1 AJO53497.1 NEVH01026112 PNF14576.1 KJ371288 KJ371289 AJO53478.1 AJO53479.1 GFDL01009943 JAV25102.1 GGMS01017491 MBY86694.1 GFXV01006673 MBW18478.1 KK856012 PTY24634.1 DQ886272 ABI52605.1 KM609496 AJQ30120.1 DS231814 EDS31763.1 KJ371286 KJ371287 AJO53476.1 AJO53477.1 FX983735 BAX07234.1 JXJN01005583 CM000363 EDX09694.1 CM002912 KMY98364.1 KMY98367.1 EZ422265 ADD18541.1 GBHO01037376 GBHO01037375 GBRD01009773 GDHC01019273 GDHC01003736 JAG06228.1 JAG06229.1 JAG56051.1 JAP99355.1 JAQ14893.1 KMY98366.1 CH902618 KPU78103.1 EDV39606.2 CH964154 EDW79922.2 AE014296 ACL83265.2 KK853393 KDR07905.1 AAF50434.1 KPU78101.1 GL447151 EFN86839.1 CM000159 KRK01013.1 AY047505 AAF50433.1 AAK77237.1 KMY98363.1 EDW93181.1 CH480815 EDW40699.1 KRK01014.1 KF647631 AHB50493.1 CH954178 KQS43455.1
BABH01024482 ODYU01011457 SOQ57199.1 NWSH01000730 PCG74547.1 RSAL01000003 RVE54841.1 JTDY01000658 KOB76299.1 AGBW02011011 OWR47392.1 GAIX01014292 JAA78268.1 AK401463 KQ459302 BAM18085.1 KPJ01762.1 KQ461175 KPJ07851.1 AJWK01027438 GFDF01001824 JAV12260.1 GFDF01001896 JAV12188.1 KQ971372 EFA09539.1 GDAI01000128 JAI17475.1 GFDG01001899 JAV16900.1 KA644621 AFP59250.1 JRES01000036 KNC34713.1 GFDG01003229 JAV15570.1 GECZ01006426 JAS63343.1 GEZM01037003 GEZM01037002 JAV82270.1 AJVK01023348 AJVK01023349 AJVK01023350 AJVK01023351 GAKP01012162 GAKP01012161 JAC46790.1 GDHF01018042 JAI34272.1 GAKP01012160 JAC46792.1 GBYB01011477 JAG81244.1 GDHF01027592 GDHF01016721 GDHF01013834 JAI24722.1 JAI35593.1 JAI38480.1 GDHF01023594 JAI28720.1 GDHF01020874 JAI31440.1 GEDC01029712 JAS07586.1 GBXI01014999 GBXI01000271 JAC99292.1 JAD14021.1 GALX01003372 JAB65094.1 KJ371291 AJO53481.1 KJ371290 AJO53480.1 PYGN01000020 PSN57464.1 KJ371266 AJO53456.1 KJ371166 KJ371167 KJ371168 KJ371169 KJ371170 KJ371171 KJ371172 KJ371173 KJ371174 KJ371175 KJ371176 KJ371177 KJ371178 KJ371179 KJ371180 KJ371181 KJ371182 KJ371183 KJ371184 KJ371185 KJ371186 KJ371187 KJ371188 KJ371190 KJ371191 KJ371192 KJ371194 KJ371195 KJ371196 KJ371198 KJ371199 KJ371200 KJ371201 KJ371202 KJ371203 KJ371204 KJ371205 KJ371207 KJ371208 KJ371209 KJ371210 KJ371211 KJ371212 KJ371213 KJ371214 KJ371215 KJ371216 KJ371217 KJ371218 KJ371219 KJ371220 KJ371221 KJ371222 KJ371224 KJ371225 KJ371226 KJ371227 KJ371228 KJ371229 KJ371230 KJ371231 KJ371232 KJ371234 KJ371235 KJ371237 KJ371240 KJ371242 KJ371243 KJ371244 KJ371245 KJ371246 KJ371247 KJ371248 KJ371249 KJ371250 KJ371251 KJ371252 KJ371253 KJ371254 KJ371255 KJ371256 KJ371257 KJ371258 KJ371259 KJ371260 KJ371261 KJ371262 KJ371263 KJ371264 KJ371265 KJ371267 KJ371268 KJ371269 KJ371271 KJ371272 KJ371273 KJ371274 KJ371275 KJ371276 KJ371277 KJ371278 KJ371279 KJ371280 KJ371281 KJ371282 KJ371283 KJ371284 KJ371285 AJO53356.1 AJO53357.1 AJO53358.1 AJO53359.1 AJO53360.1 AJO53361.1 AJO53362.1 AJO53363.1 AJO53364.1 AJO53365.1 AJO53366.1 AJO53367.1 AJO53368.1 AJO53369.1 AJO53370.1 AJO53371.1 AJO53372.1 AJO53373.1 AJO53374.1 AJO53375.1 AJO53376.1 AJO53377.1 AJO53378.1 AJO53380.1 AJO53381.1 AJO53382.1 AJO53384.1 AJO53385.1 AJO53386.1 AJO53388.1 AJO53389.1 AJO53390.1 AJO53391.1 AJO53392.1 AJO53393.1 AJO53394.1 AJO53395.1 AJO53397.1 AJO53398.1 AJO53399.1 AJO53400.1 AJO53401.1 AJO53402.1 AJO53403.1 AJO53404.1 AJO53405.1 AJO53406.1 AJO53407.1 AJO53408.1 AJO53409.1 AJO53410.1 AJO53411.1 AJO53412.1 AJO53414.1 AJO53415.1 AJO53416.1 AJO53417.1 AJO53418.1 AJO53419.1 AJO53420.1 AJO53421.1 AJO53422.1 AJO53424.1 AJO53425.1 AJO53427.1 AJO53430.1 AJO53432.1 AJO53433.1 AJO53434.1 AJO53435.1 AJO53436.1 AJO53437.1 AJO53438.1 AJO53439.1 AJO53440.1 AJO53441.1 AJO53442.1 AJO53443.1 AJO53444.1 AJO53445.1 AJO53446.1 AJO53447.1 AJO53448.1 AJO53449.1 AJO53450.1 AJO53451.1 AJO53452.1 AJO53453.1 AJO53454.1 AJO53455.1 AJO53457.1 AJO53458.1 AJO53459.1 AJO53461.1 AJO53462.1 AJO53463.1 AJO53464.1 AJO53465.1 AJO53466.1 AJO53467.1 AJO53468.1 AJO53469.1 AJO53470.1 AJO53471.1 AJO53472.1 AJO53473.1 AJO53474.1 AJO53475.1 KJ371238 KJ371239 AJO53428.1 AJO53429.1 KJ371223 AJO53413.1 GAMC01004653 GAMC01004652 JAC01904.1 KJ371206 KJ371233 KJ371236 AJO53396.1 AJO53423.1 AJO53426.1 KX147675 ARI45079.1 KJ371300 KJ371301 AJO53490.1 AJO53491.1 DQ440329 CH477200 ABF18362.1 EAT48221.1 KJ371270 AJO53460.1 KJ371297 KJ371299 AJO53487.1 AJO53489.1 KJ371189 KJ371193 KJ371197 AJO53379.1 AJO53383.1 AJO53387.1 KJ371241 AJO53431.1 KJ371308 KJ371309 KJ371310 KJ371311 AJO53498.1 AJO53499.1 AJO53500.1 AJO53501.1 GGMR01002457 MBY15076.1 KJ371292 KJ371293 KJ371294 KJ371295 AJO53482.1 AJO53483.1 AJO53484.1 AJO53485.1 KJ371313 KJ371315 AJO53503.1 AJO53505.1 KJ371296 KJ371298 AJO53486.1 AJO53488.1 KJ371302 KJ371303 KJ371304 KJ371306 AJO53492.1 AJO53493.1 AJO53494.1 AJO53496.1 KJ371312 KJ371314 AJO53502.1 AJO53504.1 JXUM01023082 JXUM01023083 JXUM01023084 JXUM01023085 JXUM01023086 JXUM01023087 KJ371305 KJ371307 AJO53495.1 AJO53497.1 NEVH01026112 PNF14576.1 KJ371288 KJ371289 AJO53478.1 AJO53479.1 GFDL01009943 JAV25102.1 GGMS01017491 MBY86694.1 GFXV01006673 MBW18478.1 KK856012 PTY24634.1 DQ886272 ABI52605.1 KM609496 AJQ30120.1 DS231814 EDS31763.1 KJ371286 KJ371287 AJO53476.1 AJO53477.1 FX983735 BAX07234.1 JXJN01005583 CM000363 EDX09694.1 CM002912 KMY98364.1 KMY98367.1 EZ422265 ADD18541.1 GBHO01037376 GBHO01037375 GBRD01009773 GDHC01019273 GDHC01003736 JAG06228.1 JAG06229.1 JAG56051.1 JAP99355.1 JAQ14893.1 KMY98366.1 CH902618 KPU78103.1 EDV39606.2 CH964154 EDW79922.2 AE014296 ACL83265.2 KK853393 KDR07905.1 AAF50434.1 KPU78101.1 GL447151 EFN86839.1 CM000159 KRK01013.1 AY047505 AAF50433.1 AAK77237.1 KMY98363.1 EDW93181.1 CH480815 EDW40699.1 KRK01014.1 KF647631 AHB50493.1 CH954178 KQS43455.1
Proteomes
UP000005204
UP000218220
UP000283053
UP000037510
UP000007151
UP000053268
+ More
UP000053240 UP000092461 UP000007266 UP000095301 UP000095300 UP000037069 UP000092462 UP000192223 UP000245037 UP000008820 UP000069940 UP000235965 UP000002320 UP000079169 UP000078200 UP000092460 UP000000304 UP000092443 UP000091820 UP000007801 UP000007798 UP000000803 UP000027135 UP000075885 UP000008237 UP000002282 UP000001292 UP000008711
UP000053240 UP000092461 UP000007266 UP000095301 UP000095300 UP000037069 UP000092462 UP000192223 UP000245037 UP000008820 UP000069940 UP000235965 UP000002320 UP000079169 UP000078200 UP000092460 UP000000304 UP000092443 UP000091820 UP000007801 UP000007798 UP000000803 UP000027135 UP000075885 UP000008237 UP000002282 UP000001292 UP000008711
Interpro
ProteinModelPortal
Q2F681
H9JBL2
A0A2H1WVV4
A0A2A4JR90
A0A3S2NRY6
A0A0L7LLS3
+ More
A0A212F0Y6 S4NRX2 I4DJJ5 A0A194QWA6 A0A1B0CTD9 A0A1L8E0K3 A0A1L8E0D2 D6X0W4 A0A0K8TU47 A0A1I8N1J5 A0A1L8EDV4 T1P7N4 A0A1I8P3Z8 A0A0L0CQS9 A0A1L8E9W3 A0A1I8P3X6 A0A1B6GLY1 A0A1Y1MCE9 A0A1B0D371 A0A034VY52 A0A0K8V5S9 A0A034VXI0 A0A0C9RE92 A0A0K8VIU0 A0A0K8UQX7 A0A1W4XI19 A0A0K8UYM3 A0A1B6C244 A0A0A1XSM3 V5GLZ3 A0A0C5CP30 A0A0C5CKF5 A0A2P8ZLT9 A0A0C5CP10 A0A0C5CKC5 A0A0C5CGB8 A0A0C5D1E6 W8BYY5 A0A0C5CNT3 A0A1W5YLL3 A0A0C5CP36 Q1HQW5 A0A0C5CKD6 A0A0C5CKU5 A0A0C5CKG7 A0A0C5CNZ5 A0A0C5D1K8 A0A2S2NE75 A0A0C5D1J6 A0A0C5D1L2 A0A0C5CP33 A0A0C5CP39 A0A0C5CKV9 A0A182H172 A0A0C5CKG6 A0A2J7PE17 A0A0C5CGG5 A0A1Q3FC67 A0A2S2R9R6 A0A2H8TZ88 A0A2R7WYM0 Q09JZ8 A0A0C5QRX3 B0VZM6 A0A1S3DMV7 A0A0C5CP27 A0A2Z5RE48 A0A1A9VYM5 A0A1B0AYA6 B4QLN4 A0A0J9RST1 A0A0J9RRM2 D3TLG3 A0A1A9XWJ2 A0A0A9WI82 A0A1A9WRA5 A0A0J9RR79 A0A0P8XU25 B3M5J6 B4N6D3 B7Z0E0 A0A067QHM4 A0A182PMH2 Q7KUB1 A0A0P9AJT6 E2BBR3 A0A0R1DVD9 Q9VSI6 A0A0J9RRC2 B4PG37 B4HJH5 A0A0R1DV11 V5SKH7 A0A0Q5U2W9
A0A212F0Y6 S4NRX2 I4DJJ5 A0A194QWA6 A0A1B0CTD9 A0A1L8E0K3 A0A1L8E0D2 D6X0W4 A0A0K8TU47 A0A1I8N1J5 A0A1L8EDV4 T1P7N4 A0A1I8P3Z8 A0A0L0CQS9 A0A1L8E9W3 A0A1I8P3X6 A0A1B6GLY1 A0A1Y1MCE9 A0A1B0D371 A0A034VY52 A0A0K8V5S9 A0A034VXI0 A0A0C9RE92 A0A0K8VIU0 A0A0K8UQX7 A0A1W4XI19 A0A0K8UYM3 A0A1B6C244 A0A0A1XSM3 V5GLZ3 A0A0C5CP30 A0A0C5CKF5 A0A2P8ZLT9 A0A0C5CP10 A0A0C5CKC5 A0A0C5CGB8 A0A0C5D1E6 W8BYY5 A0A0C5CNT3 A0A1W5YLL3 A0A0C5CP36 Q1HQW5 A0A0C5CKD6 A0A0C5CKU5 A0A0C5CKG7 A0A0C5CNZ5 A0A0C5D1K8 A0A2S2NE75 A0A0C5D1J6 A0A0C5D1L2 A0A0C5CP33 A0A0C5CP39 A0A0C5CKV9 A0A182H172 A0A0C5CKG6 A0A2J7PE17 A0A0C5CGG5 A0A1Q3FC67 A0A2S2R9R6 A0A2H8TZ88 A0A2R7WYM0 Q09JZ8 A0A0C5QRX3 B0VZM6 A0A1S3DMV7 A0A0C5CP27 A0A2Z5RE48 A0A1A9VYM5 A0A1B0AYA6 B4QLN4 A0A0J9RST1 A0A0J9RRM2 D3TLG3 A0A1A9XWJ2 A0A0A9WI82 A0A1A9WRA5 A0A0J9RR79 A0A0P8XU25 B3M5J6 B4N6D3 B7Z0E0 A0A067QHM4 A0A182PMH2 Q7KUB1 A0A0P9AJT6 E2BBR3 A0A0R1DVD9 Q9VSI6 A0A0J9RRC2 B4PG37 B4HJH5 A0A0R1DV11 V5SKH7 A0A0Q5U2W9
PDB
5YZI
E-value=0,
Score=1719
Ontologies
PATHWAY
00020
Citrate cycle (TCA cycle) - Bombyx mori (domestic silkworm)
00480 Glutathione metabolism - Bombyx mori (domestic silkworm)
01100 Metabolic pathways - Bombyx mori (domestic silkworm)
01200 Carbon metabolism - Bombyx mori (domestic silkworm)
01210 2-Oxocarboxylic acid metabolism - Bombyx mori (domestic silkworm)
01230 Biosynthesis of amino acids - Bombyx mori (domestic silkworm)
04146 Peroxisome - Bombyx mori (domestic silkworm)
00480 Glutathione metabolism - Bombyx mori (domestic silkworm)
01100 Metabolic pathways - Bombyx mori (domestic silkworm)
01200 Carbon metabolism - Bombyx mori (domestic silkworm)
01210 2-Oxocarboxylic acid metabolism - Bombyx mori (domestic silkworm)
01230 Biosynthesis of amino acids - Bombyx mori (domestic silkworm)
04146 Peroxisome - Bombyx mori (domestic silkworm)
GO
PANTHER
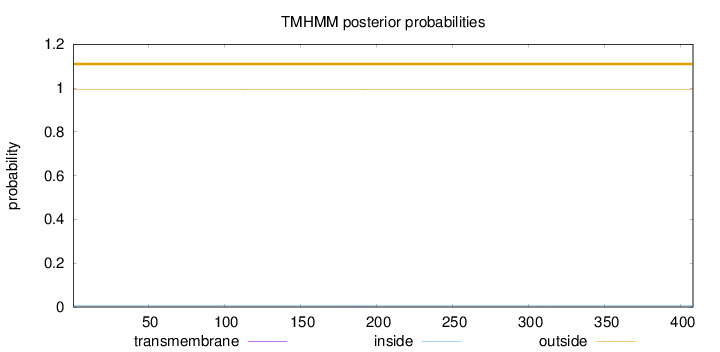
Topology
Length:
408
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
0.01489
Exp number, first 60 AAs:
0.00049
Total prob of N-in:
0.00728
outside
1 - 408
Population Genetic Test Statistics
Pi
185.356586
Theta
176.001084
Tajima's D
0.203353
CLR
0
CSRT
0.426578671066447
Interpretation
Uncertain
