Gene
KWMTBOMO05038
Pre Gene Modal
BGIBMGA012535
Annotation
PREDICTED:_desert_hedgehog_protein_B_[Papilio_machaon]
Full name
Hedgehog protein
+ More
Tiggy-winkle hedgehog protein
Tiggy-winkle hedgehog protein
Alternative Name
Sonic hedgehog protein B
Location in the cell
Mitochondrial Reliability : 1.302
Sequence
CDS
ATGCTTGCTCATGGGTGTCGGGCGGCAAGCAAGGGATGCAAGTTCAGGCTCGGTGGTGGACGTGCAGTTGAGTCGGGTTGTGGCAGTTGCTCTCAGTGTCGCTCAAGTCGCGGCGCGCGGTGGTCGTGCGTCGAGCGCGTGTGGAGTGATGTCTGCGAGATGAGGCTCTCGCTGGTGCTTCTGTGGTTGGGTGCGGCGGCGGCATGCGGCCCGGGCCGCGGTTTCAACCGTCGCCACGGACCGCGCCGCATCACCCCCCTTGTCTTTGGCCAGCACGATCCGAATGTTAGCGAAAATAAGAACACCGCCAGTGGTCCTCCCGAGGGCCGCATCACCAGAGATGACGAGAGATTTAAAGACTTAGTGCCTAATTATAATCCGGATATAGACTTTAGGGACGACGAAGGCACTGGCGCTGATCGTCTAATGACTCAGAGATGCAAGGAGAAATTAAACACTCTCGCAATCAGCGTTATGAACCAGTGGCCGGGAGTGCGGCTCCGTGTGATCGAAGGTTGGGACGAAGAGAATAATCATCTAGACAAGTCTCTCCACTACGAGGGTCGGGCTGTGGACTTAACCACCAGTGACCGAGACCGCAGCAAATACGGTATGCTGGCCCGCCTCGCTGTCGAAGCCGGCTTTGACTGGGTCTTCTATGAAAGCCGGTCTTACATTCATTGTTCTGTTAAAACAGAATCATCTGTGGGAACTGGTGCTGGCTGTTTCCCTGGCGGCTCCGTAGTTCACACTGAGAAAGGACCGAAGGATATAGCTGCCTTGCAGAAGGGAGACAAGGTCTTAGCAGCTGATGATAATGGCATGATGATTTATTCTAAGGTGCTAACATTCATCGATCGTGATCCTAATGCAACTCGACAGTTCATCGAGATAACTGCGGAGAATGGTGTTGCGATCACAACGACGCCCTCACATTTGCTCCTTCTCGCTGCAGCTGACGGTTGGCGCGATGTCTTTGCAGCTAACATCAAAGAGGGAGACGTACTATTGACAAGAGGTCGAAGTGATGTTATGAGGCCCTCGAGAGTAACGAGAACTCGTATCCTCACGAAACGAGGTGTTTTTGCCCCGCTTACCAAAGCCGGCACTATCATAGTGGATGACGTTCTGGCGTCATGCTACGCGCTAGTACGCAGTCATTCGCTTGCGCATGTTGCGATGGCACCATTGCGATGGCTTTCCAGTTGGAGCACTTCTTCAGAAACGACCAAAGGCGTCCACTGGTACGCGAGCGCGCTTTACTCTTTCGGCGACTACGTATTACCAGCATCGTATAGATATCGATAA
Protein
MLAHGCRAASKGCKFRLGGGRAVESGCGSCSQCRSSRGARWSCVERVWSDVCEMRLSLVLLWLGAAAACGPGRGFNRRHGPRRITPLVFGQHDPNVSENKNTASGPPEGRITRDDERFKDLVPNYNPDIDFRDDEGTGADRLMTQRCKEKLNTLAISVMNQWPGVRLRVIEGWDEENNHLDKSLHYEGRAVDLTTSDRDRSKYGMLARLAVEAGFDWVFYESRSYIHCSVKTESSVGTGAGCFPGGSVVHTEKGPKDIAALQKGDKVLAADDNGMMIYSKVLTFIDRDPNATRQFIEITAENGVAITTTPSHLLLLAAADGWRDVFAANIKEGDVLLTRGRSDVMRPSRVTRTRILTKRGVFAPLTKAGTIIVDDVLASCYALVRSHSLAHVAMAPLRWLSSWSTSSETTKGVHWYASALYSFGDYVLPASYRYR
Summary
Description
Intercellular signal essential for a variety of patterning events during development.
Similarity
Belongs to the hedgehog family.
Keywords
Autocatalytic cleavage
Calcium
Cell membrane
Complete proteome
Developmental protein
Hydrolase
Lipoprotein
Membrane
Metal-binding
Palmitate
Protease
Reference proteome
Secreted
Signal
Zinc
Feature
chain Hedgehog protein
Uniprot
A0A2A4JC83
A0A3S2P767
A0A158NGS9
A0A195FEJ8
A0A151XCU2
A0A195DXS7
+ More
A0A1J1HKS3 A3KNS3 A0A3L8DMY3 A0A2D0T6V3 A0A067R8L6 Q4PLX4 A0A1W5B7X1 B0S8K0 A0A3B4C6K3 W5MY62 K7J545 A0A060XSJ4 A0A3B4V456 W5K3H2 A0A3B4YMP6 A0A0B5JJZ7 Q90419 A0A2D0R5M0 A0A3B4Y7R0 Q6DBX7 A0A3R5VPV7 A0A139WM49 A0A3Q1B3K7 A2I5E3 A0A3P8UMD8 A0A3B4DSA4 A0A3B4C2C0 A0A1S3MZ68 E0V9Q6 A0A218UU38 A0A3B3DM54 A0A3P8UEE7 A0A0F8AQM8 A0A3B3SRJ0 A0A3B3SRJ5 A0A3P8U565 A0A3B4E4T1 M4A8D0 A0A3B3TCB6 A0A3B4CS27 A0A3B3TC00 A0A3N0YFI8 A0A3P8T8U1 A0A3Q0RSL2 A0A3Q1EN99 A0A3Q3Q8Q8 W5L125 A0A3Q3J1S2 A0A087YJ84 A0A3B5AJA7 A0A3B3YHY1 A0A3Q1GFL6 A0A3Q1BMS1 A0A3B4TGL1 A0A3Q3IT37 A0A3Q1JKA7 A0A3Q1DC99 A0A3P8XAG3 Q7Z264 A0A3Q3M4K9 A0A3Q3IQ44 A0A1S3P0K9 A0A1J0M5P1 A0A3B3CWC5 A0A3Q2YFQ0 B0WA40 A0A1W5AJS0 D3GA16 H3ATP8 A0A060YW61 Q2PXN1 A0A3N0XVC3 A0A2I4CCS1 A0A1S3R293 I3K2G6 A0A060YSW3 A0A2I4CB57 A0A3Q1ASU4 A0A3Q2CGS2 A0A3M0JVK2 H2M3V5 A0A3P9M4Y1 A0A1S3LY80 A0A3P8UI02 A0A3B3H8Y2 A0A3P9M3Y5 A0A1A6HVP4 Q6SCJ6 A0A3P9HQ24 A0A3P9HQ07 A0A060W0I4 G3PSE1
A0A1J1HKS3 A3KNS3 A0A3L8DMY3 A0A2D0T6V3 A0A067R8L6 Q4PLX4 A0A1W5B7X1 B0S8K0 A0A3B4C6K3 W5MY62 K7J545 A0A060XSJ4 A0A3B4V456 W5K3H2 A0A3B4YMP6 A0A0B5JJZ7 Q90419 A0A2D0R5M0 A0A3B4Y7R0 Q6DBX7 A0A3R5VPV7 A0A139WM49 A0A3Q1B3K7 A2I5E3 A0A3P8UMD8 A0A3B4DSA4 A0A3B4C2C0 A0A1S3MZ68 E0V9Q6 A0A218UU38 A0A3B3DM54 A0A3P8UEE7 A0A0F8AQM8 A0A3B3SRJ0 A0A3B3SRJ5 A0A3P8U565 A0A3B4E4T1 M4A8D0 A0A3B3TCB6 A0A3B4CS27 A0A3B3TC00 A0A3N0YFI8 A0A3P8T8U1 A0A3Q0RSL2 A0A3Q1EN99 A0A3Q3Q8Q8 W5L125 A0A3Q3J1S2 A0A087YJ84 A0A3B5AJA7 A0A3B3YHY1 A0A3Q1GFL6 A0A3Q1BMS1 A0A3B4TGL1 A0A3Q3IT37 A0A3Q1JKA7 A0A3Q1DC99 A0A3P8XAG3 Q7Z264 A0A3Q3M4K9 A0A3Q3IQ44 A0A1S3P0K9 A0A1J0M5P1 A0A3B3CWC5 A0A3Q2YFQ0 B0WA40 A0A1W5AJS0 D3GA16 H3ATP8 A0A060YW61 Q2PXN1 A0A3N0XVC3 A0A2I4CCS1 A0A1S3R293 I3K2G6 A0A060YSW3 A0A2I4CB57 A0A3Q1ASU4 A0A3Q2CGS2 A0A3M0JVK2 H2M3V5 A0A3P9M4Y1 A0A1S3LY80 A0A3P8UI02 A0A3B3H8Y2 A0A3P9M3Y5 A0A1A6HVP4 Q6SCJ6 A0A3P9HQ24 A0A3P9HQ07 A0A060W0I4 G3PSE1
Pubmed
EMBL
NWSH01002050
PCG69308.1
RSAL01000007
RVE54075.1
ADTU01015197
ADTU01015198
+ More
KQ981625 KYN39105.1 KQ982294 KYQ58196.1 KQ980107 KYN17678.1 CVRI01000009 CRK88629.1 BC133983 AAI33984.1 QOIP01000006 RLU21583.1 KK852865 KDR14772.1 DQ066428 AAY68016.1 BX942813 AHAT01034473 AAZX01000353 AAZX01013691 FR905933 CDQ82237.1 KM655588 AJG05934.1 U30710 U68237 BC078321 AAH78321.1 MH168557 QAA78721.1 KQ971317 KYB28973.1 EF100667 ABM66102.1 DS234995 EEB10091.1 MUZQ01000145 OWK56872.1 KQ041128 KKF30256.1 RJVU01044706 ROL44611.1 AYCK01016599 AYCK01016600 AY281133 AAP38181.1 KX365116 APD15679.1 DS231868 EDS40773.1 FJ603040 ACU45353.1 AFYH01119219 FR914690 CDQ93225.1 DQ296048 ABB99398.1 RJVU01059775 ROJ66287.1 AERX01016233 FR919121 CDQ94938.1 QRBI01000123 RMC04765.1 LZPO01008088 OBS82311.1 AY460200 AAR19208.1 FR904297 CDQ58100.1
KQ981625 KYN39105.1 KQ982294 KYQ58196.1 KQ980107 KYN17678.1 CVRI01000009 CRK88629.1 BC133983 AAI33984.1 QOIP01000006 RLU21583.1 KK852865 KDR14772.1 DQ066428 AAY68016.1 BX942813 AHAT01034473 AAZX01000353 AAZX01013691 FR905933 CDQ82237.1 KM655588 AJG05934.1 U30710 U68237 BC078321 AAH78321.1 MH168557 QAA78721.1 KQ971317 KYB28973.1 EF100667 ABM66102.1 DS234995 EEB10091.1 MUZQ01000145 OWK56872.1 KQ041128 KKF30256.1 RJVU01044706 ROL44611.1 AYCK01016599 AYCK01016600 AY281133 AAP38181.1 KX365116 APD15679.1 DS231868 EDS40773.1 FJ603040 ACU45353.1 AFYH01119219 FR914690 CDQ93225.1 DQ296048 ABB99398.1 RJVU01059775 ROJ66287.1 AERX01016233 FR919121 CDQ94938.1 QRBI01000123 RMC04765.1 LZPO01008088 OBS82311.1 AY460200 AAR19208.1 FR904297 CDQ58100.1
Proteomes
UP000218220
UP000283053
UP000005205
UP000078541
UP000075809
UP000078492
+ More
UP000183832 UP000279307 UP000221080 UP000027135 UP000192224 UP000000437 UP000261440 UP000018468 UP000002358 UP000193380 UP000261420 UP000018467 UP000261360 UP000007266 UP000257160 UP000265120 UP000087266 UP000009046 UP000197619 UP000261560 UP000265080 UP000261540 UP000002852 UP000261340 UP000257200 UP000261600 UP000028760 UP000261400 UP000261480 UP000265040 UP000265140 UP000261660 UP000264820 UP000002320 UP000008672 UP000192220 UP000005207 UP000265020 UP000269221 UP000001038 UP000265180 UP000092124 UP000265200 UP000007635
UP000183832 UP000279307 UP000221080 UP000027135 UP000192224 UP000000437 UP000261440 UP000018468 UP000002358 UP000193380 UP000261420 UP000018467 UP000261360 UP000007266 UP000257160 UP000265120 UP000087266 UP000009046 UP000197619 UP000261560 UP000265080 UP000261540 UP000002852 UP000261340 UP000257200 UP000261600 UP000028760 UP000261400 UP000261480 UP000265040 UP000265140 UP000261660 UP000264820 UP000002320 UP000008672 UP000192220 UP000005207 UP000265020 UP000269221 UP000001038 UP000265180 UP000092124 UP000265200 UP000007635
Interpro
Gene 3D
ProteinModelPortal
A0A2A4JC83
A0A3S2P767
A0A158NGS9
A0A195FEJ8
A0A151XCU2
A0A195DXS7
+ More
A0A1J1HKS3 A3KNS3 A0A3L8DMY3 A0A2D0T6V3 A0A067R8L6 Q4PLX4 A0A1W5B7X1 B0S8K0 A0A3B4C6K3 W5MY62 K7J545 A0A060XSJ4 A0A3B4V456 W5K3H2 A0A3B4YMP6 A0A0B5JJZ7 Q90419 A0A2D0R5M0 A0A3B4Y7R0 Q6DBX7 A0A3R5VPV7 A0A139WM49 A0A3Q1B3K7 A2I5E3 A0A3P8UMD8 A0A3B4DSA4 A0A3B4C2C0 A0A1S3MZ68 E0V9Q6 A0A218UU38 A0A3B3DM54 A0A3P8UEE7 A0A0F8AQM8 A0A3B3SRJ0 A0A3B3SRJ5 A0A3P8U565 A0A3B4E4T1 M4A8D0 A0A3B3TCB6 A0A3B4CS27 A0A3B3TC00 A0A3N0YFI8 A0A3P8T8U1 A0A3Q0RSL2 A0A3Q1EN99 A0A3Q3Q8Q8 W5L125 A0A3Q3J1S2 A0A087YJ84 A0A3B5AJA7 A0A3B3YHY1 A0A3Q1GFL6 A0A3Q1BMS1 A0A3B4TGL1 A0A3Q3IT37 A0A3Q1JKA7 A0A3Q1DC99 A0A3P8XAG3 Q7Z264 A0A3Q3M4K9 A0A3Q3IQ44 A0A1S3P0K9 A0A1J0M5P1 A0A3B3CWC5 A0A3Q2YFQ0 B0WA40 A0A1W5AJS0 D3GA16 H3ATP8 A0A060YW61 Q2PXN1 A0A3N0XVC3 A0A2I4CCS1 A0A1S3R293 I3K2G6 A0A060YSW3 A0A2I4CB57 A0A3Q1ASU4 A0A3Q2CGS2 A0A3M0JVK2 H2M3V5 A0A3P9M4Y1 A0A1S3LY80 A0A3P8UI02 A0A3B3H8Y2 A0A3P9M3Y5 A0A1A6HVP4 Q6SCJ6 A0A3P9HQ24 A0A3P9HQ07 A0A060W0I4 G3PSE1
A0A1J1HKS3 A3KNS3 A0A3L8DMY3 A0A2D0T6V3 A0A067R8L6 Q4PLX4 A0A1W5B7X1 B0S8K0 A0A3B4C6K3 W5MY62 K7J545 A0A060XSJ4 A0A3B4V456 W5K3H2 A0A3B4YMP6 A0A0B5JJZ7 Q90419 A0A2D0R5M0 A0A3B4Y7R0 Q6DBX7 A0A3R5VPV7 A0A139WM49 A0A3Q1B3K7 A2I5E3 A0A3P8UMD8 A0A3B4DSA4 A0A3B4C2C0 A0A1S3MZ68 E0V9Q6 A0A218UU38 A0A3B3DM54 A0A3P8UEE7 A0A0F8AQM8 A0A3B3SRJ0 A0A3B3SRJ5 A0A3P8U565 A0A3B4E4T1 M4A8D0 A0A3B3TCB6 A0A3B4CS27 A0A3B3TC00 A0A3N0YFI8 A0A3P8T8U1 A0A3Q0RSL2 A0A3Q1EN99 A0A3Q3Q8Q8 W5L125 A0A3Q3J1S2 A0A087YJ84 A0A3B5AJA7 A0A3B3YHY1 A0A3Q1GFL6 A0A3Q1BMS1 A0A3B4TGL1 A0A3Q3IT37 A0A3Q1JKA7 A0A3Q1DC99 A0A3P8XAG3 Q7Z264 A0A3Q3M4K9 A0A3Q3IQ44 A0A1S3P0K9 A0A1J0M5P1 A0A3B3CWC5 A0A3Q2YFQ0 B0WA40 A0A1W5AJS0 D3GA16 H3ATP8 A0A060YW61 Q2PXN1 A0A3N0XVC3 A0A2I4CCS1 A0A1S3R293 I3K2G6 A0A060YSW3 A0A2I4CB57 A0A3Q1ASU4 A0A3Q2CGS2 A0A3M0JVK2 H2M3V5 A0A3P9M4Y1 A0A1S3LY80 A0A3P8UI02 A0A3B3H8Y2 A0A3P9M3Y5 A0A1A6HVP4 Q6SCJ6 A0A3P9HQ24 A0A3P9HQ07 A0A060W0I4 G3PSE1
PDB
6DMY
E-value=3.50154e-73,
Score=699
Ontologies
GO
GO:0016539
GO:0005615
GO:0007267
GO:0005886
GO:0008233
GO:0007275
GO:0009073
GO:0046872
GO:0003856
GO:0016491
GO:0016021
GO:0001957
GO:0048794
GO:0048795
GO:0001649
GO:0005113
GO:0007224
GO:0048709
GO:0010468
GO:0005509
GO:0001708
GO:0055002
GO:0060956
GO:0048663
GO:0048703
GO:0030900
GO:0030182
GO:0048702
GO:0042063
GO:0045880
GO:0021513
GO:2000063
GO:0014858
GO:0005539
GO:0060516
GO:0048538
GO:0035116
GO:0048645
GO:0007405
GO:0045109
GO:0021978
GO:0045944
GO:0030177
GO:0046638
GO:0008209
GO:2000357
GO:0060685
GO:0048617
GO:0046639
GO:0014706
GO:0060664
GO:2000358
GO:0006897
GO:0097190
GO:0060458
GO:0014003
GO:0060428
GO:0048859
GO:0031016
GO:0051155
GO:0090370
GO:0007411
GO:0002320
GO:0060445
GO:0001755
GO:0060916
GO:0060459
GO:0001658
GO:0007596
GO:0071285
GO:0001570
GO:0048706
GO:0060523
GO:0045121
GO:0060769
GO:0014902
GO:0061189
GO:0060020
GO:0060174
GO:0043010
GO:0042733
GO:0043237
GO:0060463
GO:0060070
GO:0060783
GO:0030539
GO:0060782
GO:0002052
GO:0009986
GO:0061053
GO:0009949
GO:0033092
GO:0060662
GO:0021794
GO:0008270
GO:0032435
GO:0007228
GO:2001054
GO:1904339
GO:0030901
GO:0031012
GO:0007398
GO:0030336
GO:0030878
GO:0060447
GO:0003140
GO:0042307
GO:0030010
GO:0035115
GO:0042481
GO:0021938
GO:0034244
GO:0060484
GO:0048643
GO:0043588
GO:0048557
GO:0060439
GO:1905327
GO:0045059
GO:0060021
GO:0090090
GO:0007442
GO:0042130
GO:0043369
GO:0045445
GO:0051781
GO:0048839
GO:0034504
GO:0072136
GO:0060840
GO:0002076
GO:0021522
GO:0048714
GO:0001947
GO:0021904
GO:0045060
GO:0021930
GO:2000062
GO:0001569
GO:2000729
GO:0006508
GO:0005685
GO:0003677
GO:0005515
GO:0016742
GO:0003824
PANTHER
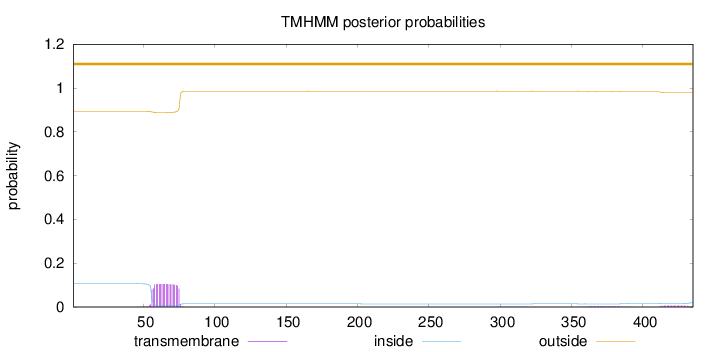
Topology
Subcellular location
Cell membrane
The N-terminal peptide remains associated with the cell surface. With evidence from 1 publications.
The C-terminal peptide diffuses from the cell. With evidence from 1 publications.
The C-terminal peptide diffuses from the cell. With evidence from 1 publications.
Length:
435
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
2.24550000000001
Exp number, first 60 AAs:
0.5049
Total prob of N-in:
0.10660
outside
1 - 435
Population Genetic Test Statistics
Pi
212.435251
Theta
184.591793
Tajima's D
0.64625
CLR
0.467407
CSRT
0.557322133893305
Interpretation
Uncertain
