Gene
KWMTBOMO01410
Pre Gene Modal
BGIBMGA012813
Annotation
endonuclease-reverse_transcriptase_[Bombyx_mori]
Location in the cell
Nuclear Reliability : 2.145
Sequence
CDS
ATGTTGGGGTCTGAGGTTTTGCGGGCCTTGAAAGCTGCTAAAACAGGTAAATCACCGGGTCCTGATAATATACCGATCGAAATACTAAAGTTGCTGGAAGAAGATCAACTGGACGCTCTCGCACAGTTCTTCAATAACATCTATGAAACTGGCAATCTTCCTAGTGACTGGCTAAAATCCACATTCATCACGATACCCAAGAAGCAAAACGCCAAAAAATCTGGAGAGTATCGTATGATTAGTCTGATGAGCCATGTTCTAAAAGTATTCTTGAACATCGTACAGAATAGAATACGGTCAAAATGTGATGAACAACTGGGCGATAGCCAATTTGGATTTCGATCTGGTGTTGGCACCAGAGAAGCTCTTTTCGCTCTCAATGTTCTCGTACAGAAATGCAGGGACATGCAAACTGATGTCTTTTTGTGCTTCACAGACTACGAAAAGGCATTTGACCGAGTTAAACATCATAACTTTTCAGCCTTTTATGTGACATTGGTCTTGATGGCTGTGATGCCAAAAGCTCTTGAAAACCTTGAAGTGAGAATAGGAATCAACGGCAGGGTCGTTAACAACTTGCGTTTTGCGGACGACACTATCCTTATTGCAGCATCGGAAGCCGATTTGCAAGCAATTGTGAATAAGGTTAATGAGTACAGCGAGGAAGCCGGTTTGTCGATAAACATAAGCAAGACCAAGTTCATGGTAGTCTCAAGAAACCCGGGTTTGAGTTCAAGCGTATATGTAGCTGGAAAACAACTAGAGCGGGTACGACAGTACAAGTATTTGGCGGCTTGGGTAAATGAAGCATGGAAATCTGATCAAGAAATCAAGATCTGGATAGAAATTGCTCGGGCCCCTTTTAATAAAATGAGGAAAGTTCTTTGCTGTCGTCAACTGAATATAAAGCTCCGAGTCAGGCTACACGGCTACATACCCGTGCGGACTCAGAAGAGGTCCTACCACCAGATGTTTATGTTTATGTTTATGTGA
Protein
MLGSEVLRALKAAKTGKSPGPDNIPIEILKLLEEDQLDALAQFFNNIYETGNLPSDWLKSTFITIPKKQNAKKSGEYRMISLMSHVLKVFLNIVQNRIRSKCDEQLGDSQFGFRSGVGTREALFALNVLVQKCRDMQTDVFLCFTDYEKAFDRVKHHNFSAFYVTLVLMAVMPKALENLEVRIGINGRVVNNLRFADDTILIAASEADLQAIVNKVNEYSEEAGLSINISKTKFMVVSRNPGLSSSVYVAGKQLERVRQYKYLAAWVNEAWKSDQEIKIWIEIARAPFNKMRKVLCCRQLNIKLRVRLHGYIPVRTQKRSYHQMFMFMFM
Summary
Uniprot
D5LB38
A0A0J7KZE8
A0A2A4JBK9
A0A2S2QAF2
W4Y5A2
H3B2Y3
+ More
A0A3B1JXQ2 A0A0A9WVQ5 A0A146LUY8 A0A2H6KKM9 A0A2B7ZVN8 A0A2J7R4S5 A0A2G8KPS5 H3ALG5 A0A2J7RJ94 A0A2J7PH94 A0A2J7RLG7 A0A2J7PQQ3 A0A2J7Q5X2 A0A2J7QCG0 A0A2J7RLC6 A0A2J7PES2 A0A2J7PL99 A0A2J7R076 A0A2J7QUF4 A0A2J7QD92 A0A2J7RDM6 A0A2J7QTX6 A0A2J7PZJ9 A0A2J7PJ38 A0A2J7RL85 A0A2J7QMH0 A0A2J7R3B7 A0A2J7Q0A4 A0A2J7Q6F7 A0A2J7QQQ3 A0A2J7PJ03 A0A2J7R4W1 A0A2J7R0K9 A0A2J7PPW0 A0A3P8TFH9 A0A2J7Q7Y8 A0A2J7QLX7 A0A2J7QPS5 A0A2J7R2Y9 A0A2J7QX53 A0A2J7RA81 A0A2J7PH80 A0A2J7PSH5 A0A2J7QV96 A0A2J7QRF9 A0A2J7RQT0 A0A2J7PQH3 A0A2J7QYI2 A0A2J7Q5X4 A0A2J7Q2Y3 A0A2J7R696 A0A2J7R8W8 A0A2J7Q647 A0A2J7R2D1 A0A2J7QSW0 A0A2J7PTS1 A0A2J7QEJ0 A0A2J7RBX3 A0A2J7PY33 A0A2J7RAC6 A0A2J7Q0P9 A0A2J7Q2T8 A0A2J7RJY3 A0A2J7PFC5 A0A2J7PHX9 A0A2J7QJ13 A0A2J7PWV5 A0A2S2QSL5 A0A2J7RBD8 A0A2J7RNM5 A0A2J7RL19 A0A2J7PMY4 A0A2J7R639 A0A2J7R7D2 A0A2J7PC89 A0A2J7QGX8 A0A2J7QLQ1 A0A2J7Q3K9 A0A2J7RES7 A0A2J7PNH4 A0A2J7QI56 A0A2J7RBL5 A0A2J7RRE4 A0A2J7PBF1 A0A2J7Q6T5 A0A2J7RPX5 A0A2J7QCG2 A0A2J7PF68 A0A2J7QSZ5 A0A2J7Q649 A0A2J7PZN7 A0A2J7Q5D5
A0A3B1JXQ2 A0A0A9WVQ5 A0A146LUY8 A0A2H6KKM9 A0A2B7ZVN8 A0A2J7R4S5 A0A2G8KPS5 H3ALG5 A0A2J7RJ94 A0A2J7PH94 A0A2J7RLG7 A0A2J7PQQ3 A0A2J7Q5X2 A0A2J7QCG0 A0A2J7RLC6 A0A2J7PES2 A0A2J7PL99 A0A2J7R076 A0A2J7QUF4 A0A2J7QD92 A0A2J7RDM6 A0A2J7QTX6 A0A2J7PZJ9 A0A2J7PJ38 A0A2J7RL85 A0A2J7QMH0 A0A2J7R3B7 A0A2J7Q0A4 A0A2J7Q6F7 A0A2J7QQQ3 A0A2J7PJ03 A0A2J7R4W1 A0A2J7R0K9 A0A2J7PPW0 A0A3P8TFH9 A0A2J7Q7Y8 A0A2J7QLX7 A0A2J7QPS5 A0A2J7R2Y9 A0A2J7QX53 A0A2J7RA81 A0A2J7PH80 A0A2J7PSH5 A0A2J7QV96 A0A2J7QRF9 A0A2J7RQT0 A0A2J7PQH3 A0A2J7QYI2 A0A2J7Q5X4 A0A2J7Q2Y3 A0A2J7R696 A0A2J7R8W8 A0A2J7Q647 A0A2J7R2D1 A0A2J7QSW0 A0A2J7PTS1 A0A2J7QEJ0 A0A2J7RBX3 A0A2J7PY33 A0A2J7RAC6 A0A2J7Q0P9 A0A2J7Q2T8 A0A2J7RJY3 A0A2J7PFC5 A0A2J7PHX9 A0A2J7QJ13 A0A2J7PWV5 A0A2S2QSL5 A0A2J7RBD8 A0A2J7RNM5 A0A2J7RL19 A0A2J7PMY4 A0A2J7R639 A0A2J7R7D2 A0A2J7PC89 A0A2J7QGX8 A0A2J7QLQ1 A0A2J7Q3K9 A0A2J7RES7 A0A2J7PNH4 A0A2J7QI56 A0A2J7RBL5 A0A2J7RRE4 A0A2J7PBF1 A0A2J7Q6T5 A0A2J7RPX5 A0A2J7QCG2 A0A2J7PF68 A0A2J7QSZ5 A0A2J7Q649 A0A2J7PZN7 A0A2J7Q5D5
EMBL
GU815089
ADF18552.1
LBMM01001656
KMQ95932.1
NWSH01001998
PCG69477.1
+ More
GGMS01004979 MBY74182.1 AAGJ04160952 AFYH01119920 GBHO01033031 GBHO01015473 GBHO01015472 GBHO01015471 GBHO01015469 GBHO01013134 GBHO01013133 GBHO01013129 GBHO01013128 JAG10573.1 JAG28131.1 JAG28132.1 JAG28133.1 JAG28135.1 JAG30470.1 JAG30471.1 JAG30475.1 JAG30476.1 GDHC01007098 GDHC01001590 JAQ11531.1 JAQ17039.1 BDSA01000081 GBE63537.1 PDHN01000994 PGH37471.1 NEVH01007402 PNF35832.1 MRZV01000437 PIK50012.1 AFYH01197936 NEVH01002994 PNF40909.1 NEVH01025142 PNF15703.1 NEVH01002684 PNF41680.1 NEVH01022635 PNF18665.1 NEVH01017543 PNF23974.1 NEVH01016289 PNF26276.1 NEVH01002690 PNF41636.1 NEVH01026089 PNF14819.1 NEVH01024426 PNF17117.1 NEVH01008283 PNF34242.1 NEVH01011186 PNF32215.1 NEVH01015826 PNF26547.1 NEVH01005288 PNF38934.1 NEVH01011192 PNF32037.1 NEVH01020333 PNF21767.1 NEVH01024952 PNF16338.1 NEVH01002692 PNF41604.1 NEVH01013208 PNF29772.1 NEVH01007822 PNF35313.1 NEVH01019987 PNF22012.1 NEVH01017478 PNF24170.1 NEVH01012083 PNF30919.1 PNF16321.1 PNF35873.1 NEVH01008232 PNF34368.1 NEVH01022644 PNF18361.1 NEVH01017000 PNF24690.1 NEVH01013241 PNF29590.1 NEVH01012088 PNF30586.1 NEVH01007824 PNF35186.1 NEVH01009398 PNF33134.1 NEVH01006576 PNF37748.1 NEVH01025149 PNF15684.1 NEVH01021931 PNF19256.1 NEVH01010478 PNF32508.1 NEVH01011898 PNF31156.1 NEVH01000618 PNF43201.1 NEVH01022636 PNF18588.1 NEVH01009090 PNF33636.1 PNF23975.1 NEVH01019075 PNF22938.1 NEVH01006936 PNF36357.1 NEVH01024428 NEVH01017533 NEVH01016946 NEVH01015820 NEVH01015300 NEVH01013960 NEVH01010477 NEVH01006721 NEVH01005894 NEVH01003749 NEVH01003737 NEVH01000598 PNF17088.1 PNF24133.1 PNF25000.1 PNF26655.1 PNF27272.1 PNF28247.1 PNF32492.1 PNF32520.1 PNF37274.1 PNF38220.1 PNF40102.1 PNF40402.1 PNF43530.1 NEVH01017541 NEVH01006570 NEVH01001345 PNF24053.1 PNF37832.1 PNF42982.1 NEVH01007834 PNF34995.1 NEVH01011217 PNF31657.1 NEVH01021221 PNF19729.1 NEVH01015309 PNF27009.1 NEVH01005889 PNF38332.1 NEVH01020850 PNF21258.1 NEVH01006574 PNF37787.1 NEVH01019964 PNF22170.1 NEVH01019077 PNF22889.1 NEVH01002982 PNF41147.1 NEVH01025668 PNF15041.1 NEVH01025130 PNF15934.1 NEVH01013563 PNF28562.1 NEVH01020867 PNF20816.1 GGMS01011307 MBY80510.1 NEVH01005906 PNF38146.1 NEVH01002150 PNF42436.1 NEVH01002700 PNF41535.1 NEVH01023960 PNF17698.1 NEVH01006981 PNF36299.1 NEVH01006734 PNF36732.1 NEVH01027067 PNF13944.1 NEVH01014359 PNF27813.1 NEVH01013243 PNF29503.1 NEVH01019066 PNF23173.1 NEVH01004944 PNF39341.1 NEVH01023953 PNF17877.1 NEVH01013959 PNF28275.1 NEVH01005896 PNF38212.1 NEVH01000609 PNF43402.1 NEVH01027104 PNF13657.1 NEVH01017452 PNF24293.1 NEVH01020851 NEVH01013954 NEVH01001358 NEVH01001354 PNF21237.1 PNF28332.1 PNF37782.1 PNF42792.1 PNF42884.1 PNF26277.1 NEVH01026085 PNF14968.1 NEVH01011205 PNF31711.1 NEVH01017540 PNF24056.1 PNF21790.1 NEVH01017570 PNF23798.1
GGMS01004979 MBY74182.1 AAGJ04160952 AFYH01119920 GBHO01033031 GBHO01015473 GBHO01015472 GBHO01015471 GBHO01015469 GBHO01013134 GBHO01013133 GBHO01013129 GBHO01013128 JAG10573.1 JAG28131.1 JAG28132.1 JAG28133.1 JAG28135.1 JAG30470.1 JAG30471.1 JAG30475.1 JAG30476.1 GDHC01007098 GDHC01001590 JAQ11531.1 JAQ17039.1 BDSA01000081 GBE63537.1 PDHN01000994 PGH37471.1 NEVH01007402 PNF35832.1 MRZV01000437 PIK50012.1 AFYH01197936 NEVH01002994 PNF40909.1 NEVH01025142 PNF15703.1 NEVH01002684 PNF41680.1 NEVH01022635 PNF18665.1 NEVH01017543 PNF23974.1 NEVH01016289 PNF26276.1 NEVH01002690 PNF41636.1 NEVH01026089 PNF14819.1 NEVH01024426 PNF17117.1 NEVH01008283 PNF34242.1 NEVH01011186 PNF32215.1 NEVH01015826 PNF26547.1 NEVH01005288 PNF38934.1 NEVH01011192 PNF32037.1 NEVH01020333 PNF21767.1 NEVH01024952 PNF16338.1 NEVH01002692 PNF41604.1 NEVH01013208 PNF29772.1 NEVH01007822 PNF35313.1 NEVH01019987 PNF22012.1 NEVH01017478 PNF24170.1 NEVH01012083 PNF30919.1 PNF16321.1 PNF35873.1 NEVH01008232 PNF34368.1 NEVH01022644 PNF18361.1 NEVH01017000 PNF24690.1 NEVH01013241 PNF29590.1 NEVH01012088 PNF30586.1 NEVH01007824 PNF35186.1 NEVH01009398 PNF33134.1 NEVH01006576 PNF37748.1 NEVH01025149 PNF15684.1 NEVH01021931 PNF19256.1 NEVH01010478 PNF32508.1 NEVH01011898 PNF31156.1 NEVH01000618 PNF43201.1 NEVH01022636 PNF18588.1 NEVH01009090 PNF33636.1 PNF23975.1 NEVH01019075 PNF22938.1 NEVH01006936 PNF36357.1 NEVH01024428 NEVH01017533 NEVH01016946 NEVH01015820 NEVH01015300 NEVH01013960 NEVH01010477 NEVH01006721 NEVH01005894 NEVH01003749 NEVH01003737 NEVH01000598 PNF17088.1 PNF24133.1 PNF25000.1 PNF26655.1 PNF27272.1 PNF28247.1 PNF32492.1 PNF32520.1 PNF37274.1 PNF38220.1 PNF40102.1 PNF40402.1 PNF43530.1 NEVH01017541 NEVH01006570 NEVH01001345 PNF24053.1 PNF37832.1 PNF42982.1 NEVH01007834 PNF34995.1 NEVH01011217 PNF31657.1 NEVH01021221 PNF19729.1 NEVH01015309 PNF27009.1 NEVH01005889 PNF38332.1 NEVH01020850 PNF21258.1 NEVH01006574 PNF37787.1 NEVH01019964 PNF22170.1 NEVH01019077 PNF22889.1 NEVH01002982 PNF41147.1 NEVH01025668 PNF15041.1 NEVH01025130 PNF15934.1 NEVH01013563 PNF28562.1 NEVH01020867 PNF20816.1 GGMS01011307 MBY80510.1 NEVH01005906 PNF38146.1 NEVH01002150 PNF42436.1 NEVH01002700 PNF41535.1 NEVH01023960 PNF17698.1 NEVH01006981 PNF36299.1 NEVH01006734 PNF36732.1 NEVH01027067 PNF13944.1 NEVH01014359 PNF27813.1 NEVH01013243 PNF29503.1 NEVH01019066 PNF23173.1 NEVH01004944 PNF39341.1 NEVH01023953 PNF17877.1 NEVH01013959 PNF28275.1 NEVH01005896 PNF38212.1 NEVH01000609 PNF43402.1 NEVH01027104 PNF13657.1 NEVH01017452 PNF24293.1 NEVH01020851 NEVH01013954 NEVH01001358 NEVH01001354 PNF21237.1 PNF28332.1 PNF37782.1 PNF42792.1 PNF42884.1 PNF26277.1 NEVH01026085 PNF14968.1 NEVH01011205 PNF31711.1 NEVH01017540 PNF24056.1 PNF21790.1 NEVH01017570 PNF23798.1
Proteomes
PRIDE
Interpro
SUPFAM
SSF56219
SSF56219
Gene 3D
ProteinModelPortal
D5LB38
A0A0J7KZE8
A0A2A4JBK9
A0A2S2QAF2
W4Y5A2
H3B2Y3
+ More
A0A3B1JXQ2 A0A0A9WVQ5 A0A146LUY8 A0A2H6KKM9 A0A2B7ZVN8 A0A2J7R4S5 A0A2G8KPS5 H3ALG5 A0A2J7RJ94 A0A2J7PH94 A0A2J7RLG7 A0A2J7PQQ3 A0A2J7Q5X2 A0A2J7QCG0 A0A2J7RLC6 A0A2J7PES2 A0A2J7PL99 A0A2J7R076 A0A2J7QUF4 A0A2J7QD92 A0A2J7RDM6 A0A2J7QTX6 A0A2J7PZJ9 A0A2J7PJ38 A0A2J7RL85 A0A2J7QMH0 A0A2J7R3B7 A0A2J7Q0A4 A0A2J7Q6F7 A0A2J7QQQ3 A0A2J7PJ03 A0A2J7R4W1 A0A2J7R0K9 A0A2J7PPW0 A0A3P8TFH9 A0A2J7Q7Y8 A0A2J7QLX7 A0A2J7QPS5 A0A2J7R2Y9 A0A2J7QX53 A0A2J7RA81 A0A2J7PH80 A0A2J7PSH5 A0A2J7QV96 A0A2J7QRF9 A0A2J7RQT0 A0A2J7PQH3 A0A2J7QYI2 A0A2J7Q5X4 A0A2J7Q2Y3 A0A2J7R696 A0A2J7R8W8 A0A2J7Q647 A0A2J7R2D1 A0A2J7QSW0 A0A2J7PTS1 A0A2J7QEJ0 A0A2J7RBX3 A0A2J7PY33 A0A2J7RAC6 A0A2J7Q0P9 A0A2J7Q2T8 A0A2J7RJY3 A0A2J7PFC5 A0A2J7PHX9 A0A2J7QJ13 A0A2J7PWV5 A0A2S2QSL5 A0A2J7RBD8 A0A2J7RNM5 A0A2J7RL19 A0A2J7PMY4 A0A2J7R639 A0A2J7R7D2 A0A2J7PC89 A0A2J7QGX8 A0A2J7QLQ1 A0A2J7Q3K9 A0A2J7RES7 A0A2J7PNH4 A0A2J7QI56 A0A2J7RBL5 A0A2J7RRE4 A0A2J7PBF1 A0A2J7Q6T5 A0A2J7RPX5 A0A2J7QCG2 A0A2J7PF68 A0A2J7QSZ5 A0A2J7Q649 A0A2J7PZN7 A0A2J7Q5D5
A0A3B1JXQ2 A0A0A9WVQ5 A0A146LUY8 A0A2H6KKM9 A0A2B7ZVN8 A0A2J7R4S5 A0A2G8KPS5 H3ALG5 A0A2J7RJ94 A0A2J7PH94 A0A2J7RLG7 A0A2J7PQQ3 A0A2J7Q5X2 A0A2J7QCG0 A0A2J7RLC6 A0A2J7PES2 A0A2J7PL99 A0A2J7R076 A0A2J7QUF4 A0A2J7QD92 A0A2J7RDM6 A0A2J7QTX6 A0A2J7PZJ9 A0A2J7PJ38 A0A2J7RL85 A0A2J7QMH0 A0A2J7R3B7 A0A2J7Q0A4 A0A2J7Q6F7 A0A2J7QQQ3 A0A2J7PJ03 A0A2J7R4W1 A0A2J7R0K9 A0A2J7PPW0 A0A3P8TFH9 A0A2J7Q7Y8 A0A2J7QLX7 A0A2J7QPS5 A0A2J7R2Y9 A0A2J7QX53 A0A2J7RA81 A0A2J7PH80 A0A2J7PSH5 A0A2J7QV96 A0A2J7QRF9 A0A2J7RQT0 A0A2J7PQH3 A0A2J7QYI2 A0A2J7Q5X4 A0A2J7Q2Y3 A0A2J7R696 A0A2J7R8W8 A0A2J7Q647 A0A2J7R2D1 A0A2J7QSW0 A0A2J7PTS1 A0A2J7QEJ0 A0A2J7RBX3 A0A2J7PY33 A0A2J7RAC6 A0A2J7Q0P9 A0A2J7Q2T8 A0A2J7RJY3 A0A2J7PFC5 A0A2J7PHX9 A0A2J7QJ13 A0A2J7PWV5 A0A2S2QSL5 A0A2J7RBD8 A0A2J7RNM5 A0A2J7RL19 A0A2J7PMY4 A0A2J7R639 A0A2J7R7D2 A0A2J7PC89 A0A2J7QGX8 A0A2J7QLQ1 A0A2J7Q3K9 A0A2J7RES7 A0A2J7PNH4 A0A2J7QI56 A0A2J7RBL5 A0A2J7RRE4 A0A2J7PBF1 A0A2J7Q6T5 A0A2J7RPX5 A0A2J7QCG2 A0A2J7PF68 A0A2J7QSZ5 A0A2J7Q649 A0A2J7PZN7 A0A2J7Q5D5
PDB
5HHL
E-value=0.00801688,
Score=91
Ontologies
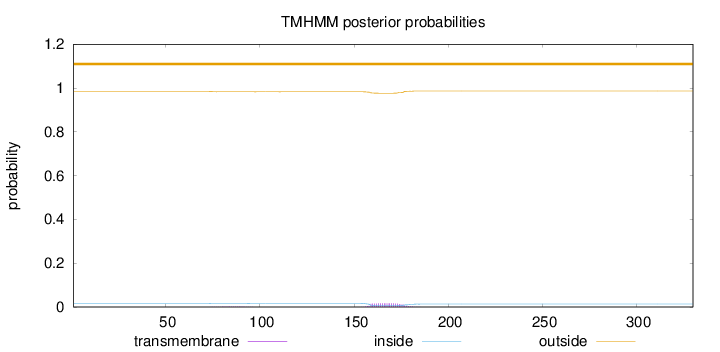
Topology
Length:
330
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
0.34024
Exp number, first 60 AAs:
0
Total prob of N-in:
0.01635
outside
1 - 330
Population Genetic Test Statistics
Pi
463.673846
Theta
202.430188
Tajima's D
4.024411
CLR
0
CSRT
0.998950052497375
Interpretation
Uncertain
