Pre Gene Modal
BGIBMGA008873
Annotation
PREDICTED:_calmodulin-like_isoform_X1_[Bombyx_mori]
Full name
Calmodulin
Location in the cell
Cytoplasmic Reliability : 2.775
Sequence
CDS
ATGGCAGTTCTCGATGCTTATAATCCTCTTCAAAATGTATCCTTACGATTCAATGAAGATCAGCTAAAAGAAATTGGCGAGTGGTTTGAGAATAATGCAAAACCTCAACTAACCAGTACTGATGGTGATCCTATCAGAGCGATAACAGTAGACAAATTCGTGTCTTTCTTAAACCTTAAAAAGTTTCATAGAAGGAATTTCCAATATCCAAGTTATGCAGAGGTAATGAATGAAGTAGAGAGATTAAAAGCTGGAGCATTACGACTATTGACTAGAGAACAAGTAATTTATATGTTGAATAAATGGGTTCTAGAACCAGACATAAAACATGAATTGTGTTTAGCTTTCAAGGTTTTCGACACAGAGAAAAGAGGTTTTCTAGAAATTGACGAACTAAAAACAATTGTAACAAGCTACGCTGAACCATTCTCGGAAGAGGAGACACGTGAAATGCTACGTGATGCAAATGTCCGTGGCGATGGAAATGTATTTTATGAAAGTTTTGTTGAAAGCCTATTCAGCGTTGCTCCTGAATTATATGAAATCAAGACAGACTATTTATACGAGGACCCTAATGAAGATCCATCGGTTCCCCCTGAGCCTGTTGTTGTAGAAGAACCTCCACCTGTACCTGTTCCTTTACCAATTAAGAAACCGAAAAACAAAAAAAAACAATAA
Protein
MAVLDAYNPLQNVSLRFNEDQLKEIGEWFENNAKPQLTSTDGDPIRAITVDKFVSFLNLKKFHRRNFQYPSYAEVMNEVERLKAGALRLLTREQVIYMLNKWVLEPDIKHELCLAFKVFDTEKRGFLEIDELKTIVTSYAEPFSEEETREMLRDANVRGDGNVFYESFVESLFSVAPELYEIKTDYLYEDPNEDPSVPPEPVVVEEPPPVPVPLPIKKPKNKKKQ
Summary
Description
Calmodulin mediates the control of a large number of enzymes, ion channels and other proteins by Ca(2+). Among the enzymes to be stimulated by the calmodulin-Ca(2+) complex are a number of protein kinases and phosphatases (By similarity).
Miscellaneous
This protein has four functional calcium-binding sites.
Similarity
Belongs to the calmodulin family.
Keywords
Calcium
Metal-binding
Repeat
Acetylation
Feature
chain Calmodulin
Uniprot
A0A2H1VVL4
A0A0L7L3D7
A0A1E7FEV9
A0A1Z5KHR0
B7GD08
A0A1Y2CK45
+ More
A0A0D3EME9 A0A0C9N183 A0A3P6APG8 L0AVQ8 A0A1X2J1V2 M4BVW6 Q4UF72 A0A1X2GUD4 A0A2H6KGU9 A0A061DEQ7 C3ZEW2 B5YMJ6 K0RWM8 A3FQ56 A0A0S4TBP9 A0A2P4Z6J1 A0A1J4ME23 F0X3V0 A0A0F7GHD6 A7AWR1 A0A1R4AB28 A0A1Y1YWW4 A0A168QGW0 Q4N4C2 A0A022QEA6 F0Y004 A0A1S8VD65 A0A0D6QT13 A0A081CP57 A0A175YNY9 S2J138 Q43447 A0A0L0HJB1 A0A024FSN6 F0W7H9 A0A163JML9 A0A2T7IQ98 A0A210PKR0 A0A0B7MXE1 A0A022RN24 A9NRI1 Q9M6U0 A0A0C9S362 Q0EEG9 C6F2P0 C6F2Q7 Q0EER8 Q5YET8 A0A2R5GGL6 A0A0L0S7G6 A0A0P1AX58 V9ET12 T0QYM7 D0N511 A0A3M6V7M3 A0A225VQN5 H3G9K1 A0A2D4CGL7 K3XBA4 A0A067CTC1 W2WPQ9 G5AIM3 A0A1W0A846 W2PZM2 B5AYD6 W2YYR9 A0A080ZWM7 A0A329SJQ3 A0A1V9YKD2 W2N0W7 A0A2P4XD10 A0A397B4W7 A0A0P5MCN7 Q71UH5 P27165 A0A0K9Q419 D8S2X6 E6ZMR7 V5E2X0 A0A077R6N2 A0A2N8UHB8 A0A0D1DU81
A0A0D3EME9 A0A0C9N183 A0A3P6APG8 L0AVQ8 A0A1X2J1V2 M4BVW6 Q4UF72 A0A1X2GUD4 A0A2H6KGU9 A0A061DEQ7 C3ZEW2 B5YMJ6 K0RWM8 A3FQ56 A0A0S4TBP9 A0A2P4Z6J1 A0A1J4ME23 F0X3V0 A0A0F7GHD6 A7AWR1 A0A1R4AB28 A0A1Y1YWW4 A0A168QGW0 Q4N4C2 A0A022QEA6 F0Y004 A0A1S8VD65 A0A0D6QT13 A0A081CP57 A0A175YNY9 S2J138 Q43447 A0A0L0HJB1 A0A024FSN6 F0W7H9 A0A163JML9 A0A2T7IQ98 A0A210PKR0 A0A0B7MXE1 A0A022RN24 A9NRI1 Q9M6U0 A0A0C9S362 Q0EEG9 C6F2P0 C6F2Q7 Q0EER8 Q5YET8 A0A2R5GGL6 A0A0L0S7G6 A0A0P1AX58 V9ET12 T0QYM7 D0N511 A0A3M6V7M3 A0A225VQN5 H3G9K1 A0A2D4CGL7 K3XBA4 A0A067CTC1 W2WPQ9 G5AIM3 A0A1W0A846 W2PZM2 B5AYD6 W2YYR9 A0A080ZWM7 A0A329SJQ3 A0A1V9YKD2 W2N0W7 A0A2P4XD10 A0A397B4W7 A0A0P5MCN7 Q71UH5 P27165 A0A0K9Q419 D8S2X6 E6ZMR7 V5E2X0 A0A077R6N2 A0A2N8UHB8 A0A0D1DU81
Pubmed
26227816
25634988
18923393
23137308
21148394
15994557
+ More
29078748 18563158 15459382 22835381 15044751 17953480 22833609 24023759 27752055 15994558 24225854 21368207 27158781 21750662 28812685 18854048 15537087 19741609 16946064 23785293 25527045 28186564 8471792 26814964 21551031 21148393 24356824 25062916 17080091
29078748 18563158 15459382 22835381 15044751 17953480 22833609 24023759 27752055 15994558 24225854 21368207 27158781 21750662 28812685 18854048 15537087 19741609 16946064 23785293 25527045 28186564 8471792 26814964 21551031 21148393 24356824 25062916 17080091
EMBL
ODYU01004705
SOQ44870.1
JTDY01003271
KOB69836.1
KV784358
OEU16674.1
+ More
BDSP01000228 GAX25622.1 CM000629 EEC43496.1 MCGO01000014 ORY47393.1 DF836488 GAN08323.1 LR031573 VDC86051.1 CP001669 AFZ79692.1 MCGE01000001 ORZ25800.1 JH597987 CR940348 UIVT01000002 UIVS01000002 CAI74267.1 SVP90739.1 SVP91263.1 MCGT01000003 ORX61610.1 BDSA01000004 GBE62230.1 LK391710 CDR97700.1 GG666612 EEN49268.1 CP001160 ACI64472.1 AGNL01026027 AGNL01000317 EJK58168.1 EJK77863.1 AAEE01000005 EAZ51365.1 LN877948 CUV04437.1 JIBK01000054 POM85609.1 LRBP01000022 OII72465.1 FX115168 FX115714 BAJ77271.1 KP283464 AKG55580.1 AAXT01000005 EDO05489.1 LN871598 SJK86197.1 MCFE01000056 ORY02522.1 AMYB01000001 OAD09219.1 AAGK01000002 EAN33001.1 KI631538 EYU26972.1 GL833122 EGB11435.1 EGB11525.1 LYON01003401 OON00159.1 GCKF01067067 JAG92885.1 DF830167 GAK68453.1 LNRQ01000008 KZM85325.1 KE124059 EPB83786.1 L19359 AAA33948.1 KQ257455 KND01103.1 FN984963 CBN20770.1 FR824075 CCA17080.1 LT554307 SAM03836.1 MACK01002631 PVC59187.1 NEDP02005595 OWF37072.1 LN722203 CEP09792.1 KI630320 EYU41429.1 EF083909 EF678300 ABK23242.1 ABR18064.1 AF150059 AAF73157.1 GCHU01015543 JAG86392.1 AB211840 BAF32140.1 EU848894 EU848895 EU848896 EU848897 EU848898 EU848899 EU848900 EU848901 EU848902 EU848903 EU848904 EU848905 EU848906 EU848907 EU848908 EU848909 EU848910 EU848922 EU848923 EU848924 EU848925 EU848926 EU848927 EU848928 ACJ77055.1 ACJ77056.1 ACJ77057.1 ACJ77058.1 ACJ77059.1 ACJ77060.1 ACJ77061.1 ACJ77062.1 ACJ77063.1 ACJ77064.1 ACJ77065.1 ACJ77066.1 ACJ77067.1 ACJ77068.1 ACJ77069.1 ACJ77070.1 ACJ77071.1 ACJ77083.1 ACJ77084.1 ACJ77085.1 ACJ77086.1 ACJ77087.1 ACJ77088.1 ACJ77089.1 EU848911 EU848912 EU848913 EU848914 EU848915 EU848916 EU848917 EU848918 EU848919 EU848920 EU848921 ACJ77072.1 ACJ77073.1 ACJ77074.1 ACJ77075.1 ACJ77076.1 ACJ77077.1 ACJ77078.1 ACJ77079.1 ACJ77080.1 ACJ77081.1 ACJ77082.1 AB211694 AB211695 AB211696 AB211697 AB211698 AB211699 AB211700 AB211701 AB211702 AB211703 AB211704 AB211705 AB211706 AB211707 AB211708 AB211709 AB211710 AB211711 AB211712 AB211713 AB211714 AB211715 AB211716 AB211717 AB211718 AB211719 AB211720 AB211721 AB211722 AB211723 AB211724 AB211725 AB211726 AB211727 AB211728 AB211729 AB211730 AB211731 AB211732 AB211733 AB211734 AB211735 AB211736 AB211737 AB211738 AB211739 AB211740 AB211741 BAF31994.1 BAF31995.1 BAF31996.1 BAF31997.1 BAF31998.1 BAF31999.1 BAF32000.1 BAF32001.1 BAF32002.1 BAF32003.1 BAF32004.1 BAF32005.1 BAF32006.1 BAF32007.1 BAF32008.1 BAF32009.1 BAF32010.1 BAF32011.1 BAF32012.1 BAF32013.1 BAF32014.1 BAF32015.1 BAF32016.1 BAF32017.1 BAF32018.1 BAF32019.1 BAF32020.1 BAF32021.1 BAF32022.1 BAF32023.1 BAF32024.1 BAF32025.1 BAF32026.1 BAF32027.1 BAF32028.1 BAF32029.1 BAF32030.1 BAF32031.1 BAF32032.1 BAF32033.1 BAF32034.1 BAF32035.1 BAF32036.1 BAF32037.1 BAF32038.1 BAF32039.1 BAF32040.1 BAF32041.1 AY542983 AAT09075.1 BEYU01000049 GBG28898.1 GG745333 KNE58371.1 CCYD01001572 CEG45313.1 ANIZ01002096 ETI42410.1 JH767138 EQC39801.1 DS028125 EEY69969.1 QLLG01000801 QKXF01000498 RMX62227.1 RQM10993.1 NBNE01003461 OWZ07635.1 DS566038 BBXB01000355 GAY04920.1 GL376575 KK583192 KDO33733.1 ANIX01002379 ETP12511.1 JH159177 EGZ04633.1 JNBS01000341 OQS06462.1 KI669598 ETN05724.1 EU880228 ACG60663.1 ANIY01002557 ETP40252.1 ANJA01002234 ETO71038.1 MJFZ01000123 RAW37013.1 JNBR01001528 OQR86126.1 KI673898 KI680603 KI693817 ETL35814.1 ETL89052.1 ETM42302.1 NCKW01011480 NCKW01005173 POM63436.1 POM73346.1 QUSZ01005233 QUTA01006111 QUTD01005103 QUTG01004770 QUTH01002336 QUTF01011279 QUTE01006986 MZMZ02002033 RHY10672.1 RHY12687.1 RHY64110.1 RHY87247.1 RHZ26519.1 RHZ29240.1 RHZ30902.1 RQM27549.1 GDIP01169555 GDIP01166044 GDIP01159824 GDIQ01161789 GDIQ01160234 GDIQ01160233 GDIP01041647 GDIP01040572 GDIP01039723 JAJ53847.1 JAK91491.1 AF085344 M83535 LFYR01000090 KMZ76016.1 GL377606 GL377600 EFJ19382.1 EFJ21170.1 FQ311432 CBQ68524.1 KI545895 EST04506.1 HG529621 CDI54632.1 LT795064 SJX64159.1 CM003150 KIS67854.1
BDSP01000228 GAX25622.1 CM000629 EEC43496.1 MCGO01000014 ORY47393.1 DF836488 GAN08323.1 LR031573 VDC86051.1 CP001669 AFZ79692.1 MCGE01000001 ORZ25800.1 JH597987 CR940348 UIVT01000002 UIVS01000002 CAI74267.1 SVP90739.1 SVP91263.1 MCGT01000003 ORX61610.1 BDSA01000004 GBE62230.1 LK391710 CDR97700.1 GG666612 EEN49268.1 CP001160 ACI64472.1 AGNL01026027 AGNL01000317 EJK58168.1 EJK77863.1 AAEE01000005 EAZ51365.1 LN877948 CUV04437.1 JIBK01000054 POM85609.1 LRBP01000022 OII72465.1 FX115168 FX115714 BAJ77271.1 KP283464 AKG55580.1 AAXT01000005 EDO05489.1 LN871598 SJK86197.1 MCFE01000056 ORY02522.1 AMYB01000001 OAD09219.1 AAGK01000002 EAN33001.1 KI631538 EYU26972.1 GL833122 EGB11435.1 EGB11525.1 LYON01003401 OON00159.1 GCKF01067067 JAG92885.1 DF830167 GAK68453.1 LNRQ01000008 KZM85325.1 KE124059 EPB83786.1 L19359 AAA33948.1 KQ257455 KND01103.1 FN984963 CBN20770.1 FR824075 CCA17080.1 LT554307 SAM03836.1 MACK01002631 PVC59187.1 NEDP02005595 OWF37072.1 LN722203 CEP09792.1 KI630320 EYU41429.1 EF083909 EF678300 ABK23242.1 ABR18064.1 AF150059 AAF73157.1 GCHU01015543 JAG86392.1 AB211840 BAF32140.1 EU848894 EU848895 EU848896 EU848897 EU848898 EU848899 EU848900 EU848901 EU848902 EU848903 EU848904 EU848905 EU848906 EU848907 EU848908 EU848909 EU848910 EU848922 EU848923 EU848924 EU848925 EU848926 EU848927 EU848928 ACJ77055.1 ACJ77056.1 ACJ77057.1 ACJ77058.1 ACJ77059.1 ACJ77060.1 ACJ77061.1 ACJ77062.1 ACJ77063.1 ACJ77064.1 ACJ77065.1 ACJ77066.1 ACJ77067.1 ACJ77068.1 ACJ77069.1 ACJ77070.1 ACJ77071.1 ACJ77083.1 ACJ77084.1 ACJ77085.1 ACJ77086.1 ACJ77087.1 ACJ77088.1 ACJ77089.1 EU848911 EU848912 EU848913 EU848914 EU848915 EU848916 EU848917 EU848918 EU848919 EU848920 EU848921 ACJ77072.1 ACJ77073.1 ACJ77074.1 ACJ77075.1 ACJ77076.1 ACJ77077.1 ACJ77078.1 ACJ77079.1 ACJ77080.1 ACJ77081.1 ACJ77082.1 AB211694 AB211695 AB211696 AB211697 AB211698 AB211699 AB211700 AB211701 AB211702 AB211703 AB211704 AB211705 AB211706 AB211707 AB211708 AB211709 AB211710 AB211711 AB211712 AB211713 AB211714 AB211715 AB211716 AB211717 AB211718 AB211719 AB211720 AB211721 AB211722 AB211723 AB211724 AB211725 AB211726 AB211727 AB211728 AB211729 AB211730 AB211731 AB211732 AB211733 AB211734 AB211735 AB211736 AB211737 AB211738 AB211739 AB211740 AB211741 BAF31994.1 BAF31995.1 BAF31996.1 BAF31997.1 BAF31998.1 BAF31999.1 BAF32000.1 BAF32001.1 BAF32002.1 BAF32003.1 BAF32004.1 BAF32005.1 BAF32006.1 BAF32007.1 BAF32008.1 BAF32009.1 BAF32010.1 BAF32011.1 BAF32012.1 BAF32013.1 BAF32014.1 BAF32015.1 BAF32016.1 BAF32017.1 BAF32018.1 BAF32019.1 BAF32020.1 BAF32021.1 BAF32022.1 BAF32023.1 BAF32024.1 BAF32025.1 BAF32026.1 BAF32027.1 BAF32028.1 BAF32029.1 BAF32030.1 BAF32031.1 BAF32032.1 BAF32033.1 BAF32034.1 BAF32035.1 BAF32036.1 BAF32037.1 BAF32038.1 BAF32039.1 BAF32040.1 BAF32041.1 AY542983 AAT09075.1 BEYU01000049 GBG28898.1 GG745333 KNE58371.1 CCYD01001572 CEG45313.1 ANIZ01002096 ETI42410.1 JH767138 EQC39801.1 DS028125 EEY69969.1 QLLG01000801 QKXF01000498 RMX62227.1 RQM10993.1 NBNE01003461 OWZ07635.1 DS566038 BBXB01000355 GAY04920.1 GL376575 KK583192 KDO33733.1 ANIX01002379 ETP12511.1 JH159177 EGZ04633.1 JNBS01000341 OQS06462.1 KI669598 ETN05724.1 EU880228 ACG60663.1 ANIY01002557 ETP40252.1 ANJA01002234 ETO71038.1 MJFZ01000123 RAW37013.1 JNBR01001528 OQR86126.1 KI673898 KI680603 KI693817 ETL35814.1 ETL89052.1 ETM42302.1 NCKW01011480 NCKW01005173 POM63436.1 POM73346.1 QUSZ01005233 QUTA01006111 QUTD01005103 QUTG01004770 QUTH01002336 QUTF01011279 QUTE01006986 MZMZ02002033 RHY10672.1 RHY12687.1 RHY64110.1 RHY87247.1 RHZ26519.1 RHZ29240.1 RHZ30902.1 RQM27549.1 GDIP01169555 GDIP01166044 GDIP01159824 GDIQ01161789 GDIQ01160234 GDIQ01160233 GDIP01041647 GDIP01040572 GDIP01039723 JAJ53847.1 JAK91491.1 AF085344 M83535 LFYR01000090 KMZ76016.1 GL377606 GL377600 EFJ19382.1 EFJ21170.1 FQ311432 CBQ68524.1 KI545895 EST04506.1 HG529621 CDI54632.1 LT795064 SJX64159.1 CM003150 KIS67854.1
Proteomes
UP000037510
UP000000759
UP000193642
UP000026960
UP000053815
UP000031512
+ More
UP000193560 UP000011713 UP000001950 UP000242146 UP000033188 UP000001554 UP000001449 UP000006726 UP000002173 UP000193498 UP000077051 UP000001949 UP000030748 UP000189906 UP000077755 UP000014254 UP000053201 UP000078561 UP000242188 UP000054107 UP000054350 UP000054928 UP000018721 UP000030762 UP000006643 UP000282087 UP000286097 UP000198211 UP000005238 UP000054177 UP000030745 UP000018958 UP000002640 UP000243217 UP000018817 UP000018948 UP000028582 UP000251314 UP000243579 UP000053864 UP000054423 UP000054532 UP000036987 UP000001514 UP000008867 UP000019377 UP000239563 UP000000561
UP000193560 UP000011713 UP000001950 UP000242146 UP000033188 UP000001554 UP000001449 UP000006726 UP000002173 UP000193498 UP000077051 UP000001949 UP000030748 UP000189906 UP000077755 UP000014254 UP000053201 UP000078561 UP000242188 UP000054107 UP000054350 UP000054928 UP000018721 UP000030762 UP000006643 UP000282087 UP000286097 UP000198211 UP000005238 UP000054177 UP000030745 UP000018958 UP000002640 UP000243217 UP000018817 UP000018948 UP000028582 UP000251314 UP000243579 UP000053864 UP000054423 UP000054532 UP000036987 UP000001514 UP000008867 UP000019377 UP000239563 UP000000561
Interpro
SUPFAM
SSF47473
SSF47473
CDD
ProteinModelPortal
A0A2H1VVL4
A0A0L7L3D7
A0A1E7FEV9
A0A1Z5KHR0
B7GD08
A0A1Y2CK45
+ More
A0A0D3EME9 A0A0C9N183 A0A3P6APG8 L0AVQ8 A0A1X2J1V2 M4BVW6 Q4UF72 A0A1X2GUD4 A0A2H6KGU9 A0A061DEQ7 C3ZEW2 B5YMJ6 K0RWM8 A3FQ56 A0A0S4TBP9 A0A2P4Z6J1 A0A1J4ME23 F0X3V0 A0A0F7GHD6 A7AWR1 A0A1R4AB28 A0A1Y1YWW4 A0A168QGW0 Q4N4C2 A0A022QEA6 F0Y004 A0A1S8VD65 A0A0D6QT13 A0A081CP57 A0A175YNY9 S2J138 Q43447 A0A0L0HJB1 A0A024FSN6 F0W7H9 A0A163JML9 A0A2T7IQ98 A0A210PKR0 A0A0B7MXE1 A0A022RN24 A9NRI1 Q9M6U0 A0A0C9S362 Q0EEG9 C6F2P0 C6F2Q7 Q0EER8 Q5YET8 A0A2R5GGL6 A0A0L0S7G6 A0A0P1AX58 V9ET12 T0QYM7 D0N511 A0A3M6V7M3 A0A225VQN5 H3G9K1 A0A2D4CGL7 K3XBA4 A0A067CTC1 W2WPQ9 G5AIM3 A0A1W0A846 W2PZM2 B5AYD6 W2YYR9 A0A080ZWM7 A0A329SJQ3 A0A1V9YKD2 W2N0W7 A0A2P4XD10 A0A397B4W7 A0A0P5MCN7 Q71UH5 P27165 A0A0K9Q419 D8S2X6 E6ZMR7 V5E2X0 A0A077R6N2 A0A2N8UHB8 A0A0D1DU81
A0A0D3EME9 A0A0C9N183 A0A3P6APG8 L0AVQ8 A0A1X2J1V2 M4BVW6 Q4UF72 A0A1X2GUD4 A0A2H6KGU9 A0A061DEQ7 C3ZEW2 B5YMJ6 K0RWM8 A3FQ56 A0A0S4TBP9 A0A2P4Z6J1 A0A1J4ME23 F0X3V0 A0A0F7GHD6 A7AWR1 A0A1R4AB28 A0A1Y1YWW4 A0A168QGW0 Q4N4C2 A0A022QEA6 F0Y004 A0A1S8VD65 A0A0D6QT13 A0A081CP57 A0A175YNY9 S2J138 Q43447 A0A0L0HJB1 A0A024FSN6 F0W7H9 A0A163JML9 A0A2T7IQ98 A0A210PKR0 A0A0B7MXE1 A0A022RN24 A9NRI1 Q9M6U0 A0A0C9S362 Q0EEG9 C6F2P0 C6F2Q7 Q0EER8 Q5YET8 A0A2R5GGL6 A0A0L0S7G6 A0A0P1AX58 V9ET12 T0QYM7 D0N511 A0A3M6V7M3 A0A225VQN5 H3G9K1 A0A2D4CGL7 K3XBA4 A0A067CTC1 W2WPQ9 G5AIM3 A0A1W0A846 W2PZM2 B5AYD6 W2YYR9 A0A080ZWM7 A0A329SJQ3 A0A1V9YKD2 W2N0W7 A0A2P4XD10 A0A397B4W7 A0A0P5MCN7 Q71UH5 P27165 A0A0K9Q419 D8S2X6 E6ZMR7 V5E2X0 A0A077R6N2 A0A2N8UHB8 A0A0D1DU81
PDB
3EKH
E-value=2.23265e-13,
Score=180
Ontologies
PATHWAY
GO
PANTHER
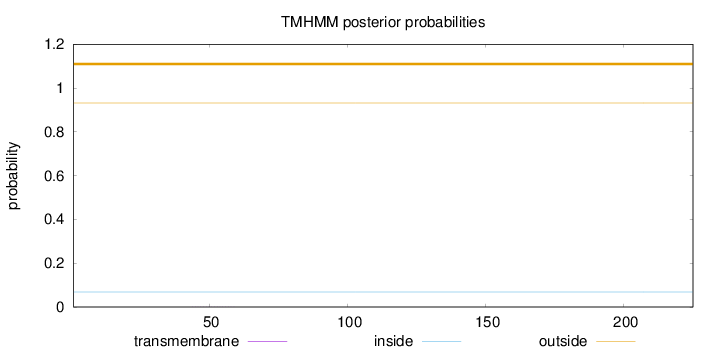
Topology
Length:
225
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
0.00349
Exp number, first 60 AAs:
0.00287
Total prob of N-in:
0.06819
outside
1 - 225
Population Genetic Test Statistics
Pi
363.088575
Theta
224.110728
Tajima's D
1.919965
CLR
0.008769
CSRT
0.867656617169142
Interpretation
Uncertain
