Gene
KWMTBOMO01001 Validated by peptides from experiments
Pre Gene Modal
BGIBMGA001347
Annotation
fibroin_P25_[Bombyx_mandarina]
Full name
Fibrohexamerin
Alternative Name
25 kDa silk glycoprotein
p25
p25
Location in the cell
Extracellular Reliability : 2.257 PlasmaMembrane Reliability : 1.766
Sequence
CDS
ATGCTGGCGCGGTGTCTAGCTGTAGCCGCTGTGGCAGTTTTGGCTTCTGCAGGGCCGCCGAGTCCGATCTACAGGCCCTGCTACTTGGACGATTACAAATGCATCTCCGACCACCTGGCCGCGATCTCGAAATGTATCCCGGGCAGAGGGCAGATCCCCTCGCAGTACGAGATACCGGTGTTCCAGTTTGAAATACCATACTTCAACGCCACCTACGTCGACCATAATCTCATTACCCGTAACCACGACAAATGCCGTGTCAGCGAATTTTATGACAATGTGAGAACTTTAAAAACAGTTTTAACCGTGGACTGTCCGTGGCTGAACTTTGAATCCAATCGGACCTTGGCTCAGCACATGTCGTTCAAGGAAGACGTCGTCTTAAGCTTCTACATCAATGGATCTTATCCTTTGATCAGGCTAACGACAGTGTTTGATAAGGGCAACAATTTCGACTTGTGTTCGGCTTTCACGTTCGCAGACCTGGCCGGGGGTCTGCCCATCTTCCACATTAACCCTAATGACCAACGCACAGCGCAGTGGTTGTCTAAAGATCTGACCCTGCTGCACATTTACGAGAGAGAGCACATCTTCGGAAAGAGGAACTGGCTGGCGAGGTCCTTCATCAGCAGAACACTCTGCGACTTCGGTTGTCACCACTGA
Protein
MLARCLAVAAVAVLASAGPPSPIYRPCYLDDYKCISDHLAAISKCIPGRGQIPSQYEIPVFQFEIPYFNATYVDHNLITRNHDKCRVSEFYDNVRTLKTVLTVDCPWLNFESNRTLAQHMSFKEDVVLSFYINGSYPLIRLTTVFDKGNNFDLCSAFTFADLAGGLPIFHINPNDQRTAQWLSKDLTLLHIYEREHIFGKRNWLARSFISRTLCDFGCHH
Summary
Subunit
Silk fibroin elementary unit consists in a disulfide-linked heavy and light chain and a p25 glycoprotein in molar ratios of 6:6:1. This results in a complex of approximately 2.3 MDa.
Miscellaneous
It is unclear whether the N-terminal residue of the mature protein is Ala-17 or Gly-18.
Keywords
Complete proteome
Direct protein sequencing
Disulfide bond
Glycoprotein
Reference proteome
Secreted
Signal
Silk protein
Feature
chain Fibrohexamerin
Uniprot
Pubmed
EMBL
BABH01038094
BABH01038095
AB001821
BAB39500.1
X04226
BP122961
+ More
BP126317 BP177743 BAAB01057839 AGBW02013452 OWR43406.1 AF009827 AF009677 GQ901976 ACX50393.1 AB001823 BAB39502.1 KQ459593 KPI96677.1 AB001825 BAB39504.1 KQ460854 KPJ11963.1 NWSH01001474 PCG71139.1 KY792994 ARE31005.1 AB195976 BAE97692.1 ODYU01003822 SOQ43056.1 JTDY01000571 KOB76636.1 NWSH01003760 PCG65847.1 KZ149916 PZC77940.1 JTDY01003497 KOB69467.1 PCG65848.1 BABH01022225 NWSH01001990 PCG69503.1 KOB69466.1 PZC77938.1 LC001864 BAS31052.1 LC001863 BAS31051.1
BP126317 BP177743 BAAB01057839 AGBW02013452 OWR43406.1 AF009827 AF009677 GQ901976 ACX50393.1 AB001823 BAB39502.1 KQ459593 KPI96677.1 AB001825 BAB39504.1 KQ460854 KPJ11963.1 NWSH01001474 PCG71139.1 KY792994 ARE31005.1 AB195976 BAE97692.1 ODYU01003822 SOQ43056.1 JTDY01000571 KOB76636.1 NWSH01003760 PCG65847.1 KZ149916 PZC77940.1 JTDY01003497 KOB69467.1 PCG65848.1 BABH01022225 NWSH01001990 PCG69503.1 KOB69466.1 PZC77938.1 LC001864 BAS31052.1 LC001863 BAS31051.1
Proteomes
Pfam
PF07294 Fibroin_P25
SUPFAM
SSF48371
SSF48371
Gene 3D
ProteinModelPortal
Ontologies
GO
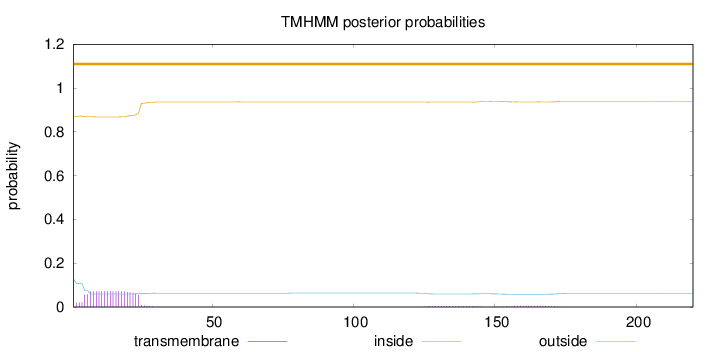
Topology
Subcellular location
Secreted
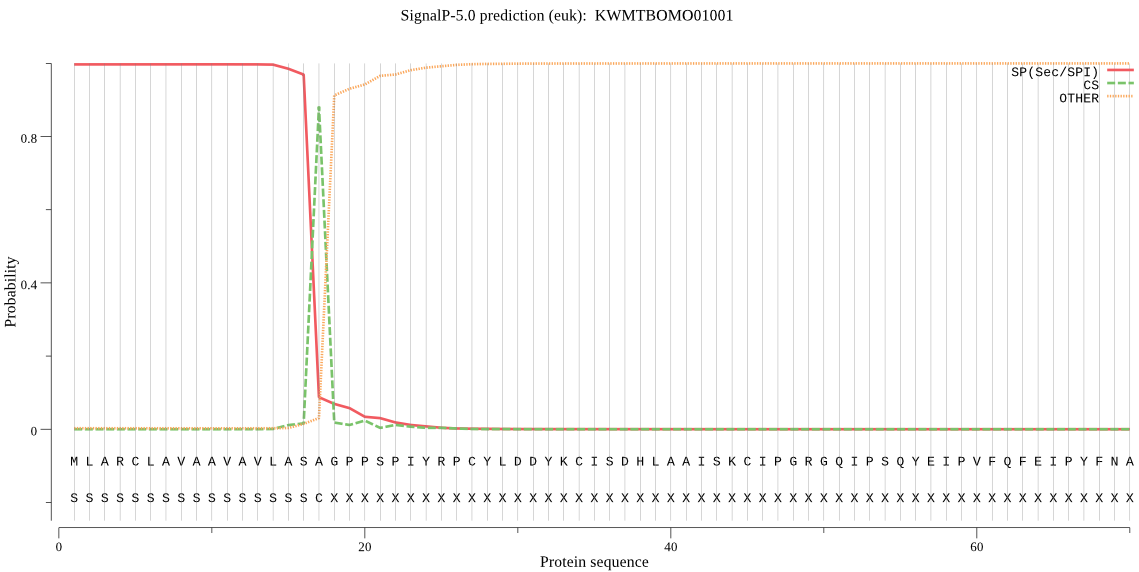
SignalP
Position: 1 - 17,
Likelihood: 0.997161
Length:
220
Number of predicted TMHs:
0
Exp number of AAs in TMHs:
1.63405
Exp number, first 60 AAs:
1.45424
Total prob of N-in:
0.12829
outside
1 - 220
Population Genetic Test Statistics
Pi
288.442107
Theta
210.220679
Tajima's D
0.910725
CLR
0.737083
CSRT
0.634218289085546
Interpretation
Uncertain
